Hydroponic Culturein Sample Preparation of Tea Shoots for RNA Sequencing
-
摘要: 为充分验证水培枝条法取样在茶树芽叶转录组测序中的可行性,将水培枝条取样法与干冰取样法所取样本提取的茶树芽叶总RNA完整性、纯度、浓度、转录组测序文库以及转录组测序结果进行比较,结果表明,水培枝条取样法与干冰取样法所取样本提取的茶树芽叶总RNA完整性均良好,纯度均较高,浓度无显著差异;2种取样方法所取样本提取的总RNA品质均符合转录组测序要求,均能成功获取效果相当的转录组测序文库以及转录组测序结果。试验表明水培枝条取样具有简单、方便的特点,更适合野外取样。Abstract: Valid sampling is essential to obtain reliable transcripts of genetic information for biological studies. For RNA sequencing, the integrity, purity and concentration of total RNA as well as the cDNA libraries and transcriptome sequencingon two specimens of tea shoots sampled by using two methods were compared. One of the methods was hydroponic culture (SM1), and another, dry ice freezing (SM2). The results showed that both of them yielded RNA extractions with high integrity and purity without significant differences on the measured concentrations. They were well satisfied for the subsequent transcriptome sequencing, also capable of obtaining the equivalent cDNA libraries and RNA sequencing. However, SM1 was simpler and more convenient than SM2 in handling and operation, and particularly suitable for field sampling. Aside from being used on tea, SM1 could be applied on other crops in preparing samples for extracting RNA as well.
-
Key words:
- tea plant (Camellia sinensis) /
- RNA sequencing /
- RNA extraction /
- hydroponic culture
-
表 1 总RNA纯度与浓度检测结果
Table 1. Purity and concentration of total RNA
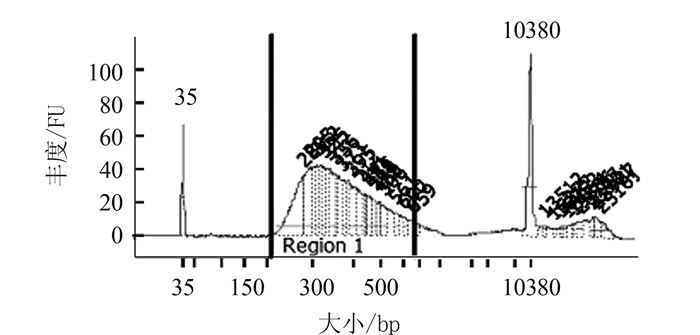
样品 A260 A280 A260/A280 质量浓度/(ng·μL-1) 总量/μg 对照样 5.30±0.21A 2.26±0.10A 2.34±0.01A 212.71±8.01A 21.27±0.80A 处理样 4.93±0.78A 2.01±0.33A 2.35±0.01A 197.72±31.38A 19.77±3.14A 注:同列数据后不同大写字母表示在0.05水平上差异显著。 表 2 对照样cDNA文库检测结果
Table 2. cDNA library of control sample
Region1区域峰值序号 大小/bp 质量浓度/(pg·μL-1) 摩尔浓度/(pmol·L-1 ) 1 280 198.12 1073.0 2 295 113.03 580.5 3 312 86.30 419.7 4 325 70.83 330.0 5 342 66.91 296.6 6 356 46.68 198.7 7 373 55.95 227.2 8 395 56.13 215.2 9 412 58.2 213.9 10 441 25.34 87.0 11 455 26.54 88.3 12 468 28.78 93.2 13 488 22.12 68.7 14 506 32.6 97.7 15 561 16.47 44.4 16 600 7.49 18.9 17 659 10.11 23.3 表 3 处理样cDNA文库检测结果
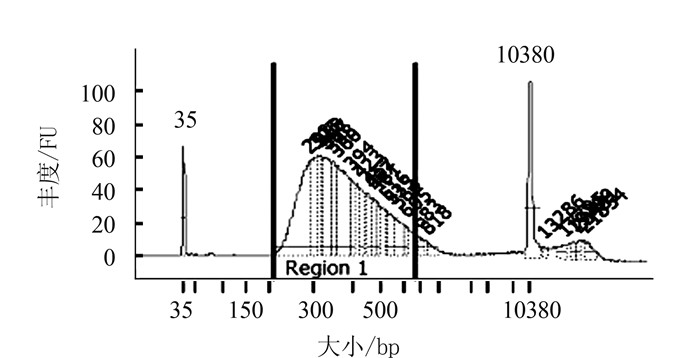
Table 3. cDNA library of tested samples
Region1区域峰值序号 大小/bp 质量浓度/(pg·μL-1) 摩尔浓度/(pmol·L-1 ) 1 292 370.99 1928.3 2 305 154.13 765.7 3 318 68.43 326.3 4 327 169.72 785.4 5 348 90.71 394.9 6 360 228.57 963.1 7 394 78.67 302.4 8 423 61.97 221.8 9 452 35.43 118.7 10 467 41.39 134.4 11 481 49.84 156.9 12 516 29.95 88.0 13 535 39.93 113.1 14 564 45.24 121.4 15 605 17.29 43.3 16 683 19.29 42.8 17 818 11.88 22.0 表 4 转录组clean reads统计结果
Table 4. Statistics of clean reads on transcriptome
统计项目 处理 对照 reads平均长度/bp 140.27 137.39 测序reads数/million 57.05 67.80 总碱基数量/Gb 8.00 9.32 测序错误率小于1%的碱基数量/% 98.95 98.62 GC含量/% 44.13 43.78 测序无法判定的碱基/% 0.00 0.00 表 5 转录组测序组装结果
Table 5. Assembly of transcriptome sequencing
统计项目 Contigs Unigenes 处理 对照 处理 对照 数量/million 6.17 6.22 0.22 0.25 总长度/million 377.63 386.81 135.49 140.23 最小长度/bp 25.00 26.00 201.00 203.00 最大长度/bp 24143.00 24143.00 24144.00 24144.00 平均长度/bp 61.18 62.36 622.79 628.11 GC含量/% 41.26 41.48 39.27 39.62 N50/bp 49.00 51.00 906.00 917.00 注:N50为将所有Contigs或Unigenes从大到小排列,从第1条序列开始累计相加,达到总长的1/2时对应的Contigs或Unigenes的长度。 表 6 不同表达水平区间的基因数量
Table 6. Number of genes between expression levels
FPKM值区间 处理 对照 数量/个 百分比/% 数量/个 百分比/% 0~1 155501 71.48 149121 68.54 1~3 32064 14.74 36905 16.96 3~15 20256 9.31 21659 9.96 15~60 7826 3.60 7971 3.66 ﹥60 1905 0.88 1896 0.87 -
[1] SHEEJA T E, DEEPA K, SANTHI R, et al. Comparative Transcriptome Analysis of Two Species of Curcuma Contrasting in a High-Value Compound Curcumin: Insights into Genetic Basis and Regulation of Biosynthesis[J]. Plant Mol Biol Rep, 2015, 33: 1825-1836. doi: 10.1007/s11105-015-0878-6 [2] QIANG ZHOU, DONG LUO, LICHAO MA, et al. Development and cross-species transferability of EST-SSR markers in Siberian wildrye (Elymus sibiricus L.) using Illumina sequencing[J]. Scientific Reports, 2016, 6: 20549. doi: 10.1038/srep20549 [3] XIAOHUI ZHANG, TONGJIN LIU, MENGMENG DUAN, et al. De novo Transcriptome Analysis of Sinapis alba in Revealing the Glucosinolate and Phytochelatin Pathways[J].Frontiers in Plant Science, 2016, 7: 259. https://www.researchgate.net/publication/297604244_De_novo_Transcriptome_Analysis_of_Sinapis_alba_in_Revealing_the_Glucosinolate_and_Phytochelatin_Pathways [4] DAN WANG, CHUN-FANG LI, CHUN-LEI MA, et al. Novel insights into the molecular mechanisms underlying the resistance of Camellia sinensis to Ectropis oblique provided by strategic transcriptomic comparisons[J]. Scientia Horticulturae, 2015, 192: 429-440. doi: 10.1016/j.scienta.2015.06.005 [5] CHUN-FANG LI, YAN ZHU, YAO YU, et al. Global transcriptome and gene regulation network for secondary metabolite biosynthesis of tea plant (Camellia sinensis)[J]. BMC Genomics, 2015, 16: 560. doi: 10.1186/s12864-015-1773-0 [6] QUANJIN WU, ZHIDAN CHEN, WEIJIANG SUN, et al. De novo Sequencing of the Leaf Transcriptome Reveals Complex Light-Responsive Regulatory Networks in Camellia sinensis cv.Baijiguan[J]. Frontiers in Plant Science, 2016, 7: 332. http://www.pubfacts.com/detail/27047513/De-novo-Sequencing-of-the-Leaf-Transcriptome-Reveals-Complex-Light-Responsive-Regulatory-Networks-in [7] 宛晓春, 夏涛.茶树次生代谢[M].北京:科学出版社, 2015. [8] SCHNEIDERBAUER A, SANDERMANN H J, ERNST D. Isolation of functional RNA from plant tissues rich in phenolic compounds[J]. Anal Biochem, 1991, 197: 91-95. doi: 10.1016/0003-2697(91)90360-6 [9] WANG C S, VODKIN L O. Extraction of RNA from tissues containing high levels of procyanidins that bind RNA[J]. Plant Mol Biol Rep, 1994, 12: 132-145. doi: 10.1007/BF02668374 [10] LEWINSOHN E, STEELE C L, CROTEAU R. Simple isolation of functional RNA from woody stems of gym nosperms[J]. Plant Mol Biol Rep, 1994, 12: 20-25. doi: 10.1007/BF02668660 [11] GRAHAM G C. A method for extraction of total RNA from Pinus radiata and other conifers[J]. Plant Mol Biol Rept, 1993, 11:32-37. doi: 10.1007/BF02670557 [12] 江昌俊, 王朝霞, 李叶云.茶树中提纯总RNA的研究[J].茶叶科学, 2000, 20(1): 27-29. http://www.cnki.com.cn/Article/CJFDTOTAL-CYKK200001005.htm [13] 林金科, 开国银.茶树RNA的提纯与鉴定[J].福建农林大学学报:自然科学版, 2003, 32(1): 70-73. http://www.cnki.com.cn/Article/CJFDTOTAL-FJND200301017.htm [14] 史成颖, 宛晓春, 江昌俊, 等.提取高质量茶树总RNA的方法研究[J].安徽农业大学学报, 2007, 34(3): 360-363. http://www.cnki.com.cn/Article/CJFDTOTAL-ANHU200703012.htm [15] 杨冬青, 王云生, 孙美莲, 等.茶树不同器官组织总RNA提取方法的研究[J].激光生物学报, 2011, 20(1): 108-115. http://www.cnki.com.cn/Article/CJFDTOTAL-JGSW201101023.htm [16] GRABHERR M G, HAAS B J, YASSOUR M, et al. Full-length transcriptome assembly from RNA-Seq data without a reference genome[J]. Nat Biotechnol, 2011, 29: 644-652. doi: 10.1038/nbt.1883 [17] LI B, DEWEY C N.RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome[J]. BMC Bioinf, 2011, 12: 323. doi: 10.1186/1471-2105-12-323 [18] 林德光.析因试验中交互作用分析的SAS技术[J].工科数学, 2001, 17(4):48-54. http://www.cnki.com.cn/Article/CJFDTOTAL-GKSX200104012.htm [19] 王镜岩, 朱圣庚, 徐长发.生物化学:第三版[M].北京:高等教育出版社, 2002. [20] ZHANG J D, SCHINDLER T, KUNG E, et al. Highly sensitive amplicon-based transcript quantification by semiconductor sequencing[J]. BMC Genomics, 2014, 15: 565. doi: 10.1186/1471-2164-15-565 [21] WANG Z, GERSTEIN M, SNYDER M. RNA-Seq: a revolutionary tool for transcriptomics[J]. Nat Rev Genet, 2009, 10: 57-63. doi: 10.1038/nrg2484 [22] 李宏, 王新力.植物组织RNA提取的难点及对策[J].生物技术通报, 1999, (1):36-39. http://www.cnki.com.cn/Article/CJFDTOTAL-SWJT901.008.htm -







 下载:
下载: