Study on Transcriptome of Moringa oleifera Lam. Leaves Based on RNA-seq
-
摘要: 为探讨辣木叶营养价值形成的遗传基础,采用RNA-seq对不同发育时期的辣木叶混合样品进行转录组测序,获得44 056 449个reads,共组装得到75 452条unigene,平均长度为795.69 nt,其中有15 824条unigene长度大于1 000 nt。采用公共数据库进行同源比对,共有30 847条unigene得到注释。其中,有18 277条unigenes被注释到GO数据库的52个功能组,9 689条unigene在COG数据库中得到注释。有10 130条unigene被富集到129条KEGG途径,包括碳代谢、氨基酸生物合成和氧化磷酸化途径。Abstract: To explore the genetic basis for the nutritive value of Moringa oleifera leaves, the leaves at different developmental stages were mixed and used for transcriptome sequencing in the present study. With RNA-seq, 44 056 449 reads were generated, then assembled into 75 452 unigenes with an average length of 795.69 nt. 15 824 of these unigenes were longer than 1 kb. 30 847 unigenes were annotated by a similarity search against public databases. Of these, 18 277 unigenes were assigned into 52 GO function groups and 9 689 unigenes were annotated by COG database. 10 130 unigenes were enriched into 129 KEGG pathways, including carbon metabolism, biosynthesis of amino acids and oxidative phosphorylation.
-
Key words:
- Moringa oleifera /
- transcriptome /
- function annotation
-
表 1 辣木叶转录组组装结果
Table 1. Summary of sequencing and de novo assembly of Moringa oleifera Lam. leaves
长度区间/nt 转录本/条 unigenes/条 200~300 30299(13.53%) 25994(34.45%) 300~500 26316(11.75%) 19970(26.47%) 500~1000 28954(12.93%) 13664(18.11%) 1000~2000 46764(20.88%) 8266(10.96%) 2000+ 91650(40.92%) 7558(10.02%) 总数量 223983 75452 总长度 438510672 60036488 N50长度 3096 1543 平均长度 1957.79 795.69 注:括号内数据为各长度区间对应的转录本、unigenes组装比例。 表 2 辣木叶unigene注释结果
Table 2. Summary of functional annotations for the assembled unigenes of Moringa oleifera Lam. leaves
数据库 注释基因
数量/条注释基因
比例/%300 nt≤长度
<1000 nt注释
基因数量/条长度≥1000 nt
注释基因
数量/条Nr 28336 37.56 9441 13533 Swiss-Prot 16454 21.81 4690 9638 GO 18277 24.22 5936 9029 COG 9689 12.84 2573 5325 KEGG 10130 13.43 3146 5288 KOG 16850 22.33 5050 8305 Pfam 19541 25.90 5323 11339 eggNOG 28948 38.37 9255 13349 All 30847 40.88 10153 13571 表 3 辣木叶转录组unigene的GO功能分类
Table 3. GO classifications of unigenes for transcriptome of Moringa oleifera Lam. leaves
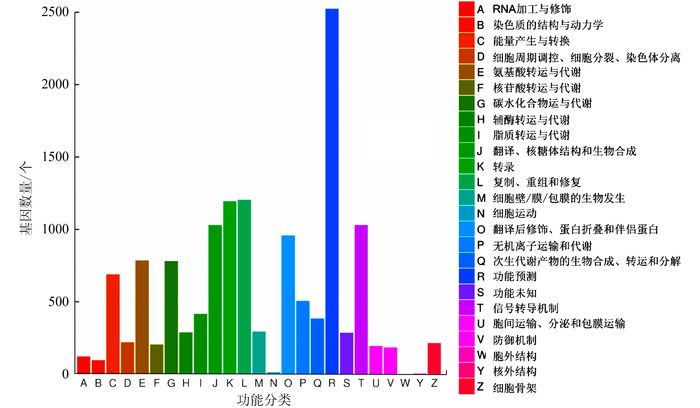
生物学过程 细胞组分 分子功能 基因功能 基因数量/个 基因功能 基因数量/个 基因功能 基因数量/个 生殖 265 胞外区 512 蛋白质结合转录因子活性 40 免疫系统过程 254 细胞 8160 核酸结合转录因子活性 500 代谢过程 12587 类核 48 催化活性 9878 细胞过程 10821 膜 4251 受体活性 89 生殖过程 856 病毒体 5 鸟嘌呤核苷酸交换因子活性 46 生物附着 36 细胞连接 293 结构分子活性 650 信号传递 932 胞外基质 7 转运体活性 1296 多细胞组织进程 1495 膜腔 535 结合 9483 发育过程 1602 大分子复合物 2218 电子载体活性 364 生长 306 胞外区 6046 抗氧化活性 145 移动 10 胞外基质组分 1 金属伴侣蛋白活性 4 单组织过程 8955 胞外区域部分 8 酶调节活性 189 生物相 33 细胞器部分 2523 蛋白标签 1 韵律过程 37 病毒体部分 5 翻译调控活性 4 刺激应答 3287 膜部分 2187 营养库活性 20 定位 2855 细胞部分 8160 分子转换器活性 244 多生物过程 759 共质体 292 生物条件 3263 生物组分组织或生物合成 2133 表 4 辣木叶转录组unigene的KEGG功能分类
Table 4. KEGG classifications of unigenes for transcriptome of Moringa oleifera Lam. leaves
代谢通路 基因数量
/个代谢通路 基因数量
/个代谢通路 基因数量
/个核糖体 517 磷酸肌醇代谢 77 缬氨酸, 亮氨酸和异亮氨酸代谢 34 碳代谢 458 磷脂酰肌醇信号系统 77 叶酸-碳单位循环代谢通路 32 氨基酸生物合成 399 核苷酸切除修复 67 类黄酮生物合成 30 内质网蛋白加工 331 苯丙氨酸,酪氨酸和色氨酸生物合成 64 硒化合物代谢 29 氧化磷酸化 285 β-丙氨酸代谢 63 其他多糖降解 29 剪接体 266 抗坏血酸代谢 62 组氨酸代谢 28 淀粉和蔗糖代谢 255 脂肪酸降解 62 磷酸肌醇生物合成 25 RNA转运 250 萜类骨架生物合成 62 高脚类,二烯类和姜素生物合成 24 糖酵解和糖异生 216 氰基氨基酸代谢 61 花生四烯酸代谢 23 嘌呤代谢 211 ABC转运体 61 硫胺代谢 22 植物激素信号转导 206 RNA聚合酶 61 核黄素代谢 21 内吞作用 197 酪氨酸代谢 60 二萜生物合成 21 mRNA监控通路 195 同源重组 60 柠檬烯、蒎烯降解 20 氨基糖和核苷酸糖代谢 183 脂肪酸生物合成 57 牛磺酸和亚牛磺酸代谢 18 植物病原菌互作 183 丙酸代谢 57 烟酸和烟酰胺代谢 18 吞噬体 176 蛋白质输出 57 生物素代谢 18 泛素介导的蛋白水解 174 色氨酸代谢 55 叶酸生物合成 18 RNA降解 162 DNA复制 55 硫中继系统 18 丙酮酸代谢 160 卟啉和叶绿素代谢 53 维生素B6代谢 16 嘧啶代谢 153 囊泡运输中的陷阱相互作用 52 赖氨酸生物合成 15 苯丙素生物合成 153 不饱和脂肪酸生物合成 51 光合作用-天线蛋白 14 半胱氨酸和蛋氨酸代谢 152 基底转录因子 50 糖胺聚糖降解 14 柠檬酸循环 139 N-多糖生物合成 48 亚油酸代谢 14 光合生物中的碳固定 129 氮代谢 48 玉米素生物合成 14 苯丙氨酸代谢 125 赖氨酸降解 47 酮体合成和降解 13 真核生物核糖体合成 123 α-亚麻酸代谢 47 糖鞘脂类生物合成-globo系列 12 二羧酸代谢 120 碱基切除修复 47 C5分支的二元酸代谢 12 精氨酸和脯氨酸代谢 117 硫代谢 46 芳香化合物降解 11 2-氧代羧酸代谢 116 光合作用 44 倍半萜和三萜类化合物生物合成 10 脂肪酸代谢 115 丁酸代谢 44 芥子油苷生物合成 10 甘油磷脂代谢 107 错配修复 44 油菜素内酯生物合成 9 丙氨酸,阿斯帕特酸和谷氨酸酯代谢 104 脂肪酸延伸 42 非同源性末端接合 9 蛋白酶体 104 自噬调节 41 其他类型o-聚糖生物合成 8 戊糖、葡萄糖醛酸转换 103 类固醇生物合成 40 单萜类生物合成 8 氨酰-tRNA生物合成 103 泛醌和其他萜类生物合成 40 鞘糖脂生物合成 6 过氧物酶体 103 醚脂类代谢 40 咖啡因代谢 5 谷胱甘肽代谢 96 哌啶和吡啶生物碱生物合成 40 花色苷生物合成 4 甘氨酸,丝氨酸和三氨酸代谢 92 植物昼夜节律 40 硫辛酸代谢 3 磷酸戊糖途径 91 泛酸盐和辅酶a生物合成 38 异黄酮生物合成 3 果糖和甘露糖代谢 91 异喹啉生物碱生物合成 37 万古霉素抗药性 3 半乳糖代谢 89 角质,亚氨酸和蜡生物合成 36 黄酮和黄酮醇生物合成 2 缬氨酸, 亮氨酸和异亮氨酸降解 81 鞘脂类代谢 36 碳青霉烯生物合成 1 甘油酯代谢 77 类胡萝卜素生物合成 36 酮化合物糖单位生物合成 1 表 5 辣木叶转录组中表达量最高的10条基因
Table 5. The 10 most abundant unigenes in transcriptome of Moringa oleifera Lam. leaves
编号 基因名称 FPKM值 c18407.graph_c0 二磷酸核酮糖羧化酶小链 15602.35 c30652.graph_c0 叶绿体叶绿素a/b结合蛋白 12537.82 c35268.graph_c0 二磷酸核酮糖羧化酶小链 8535.69 c33703.graph_c0 类Win蛋白 5152.12 c8930.graph_c0 二磷酸核酮糖羧化酶/加氧酶活化酶 3702.24 c23488.graph_c0 泛素蛋白 2702.34 c30638.graph_c1 氨基环丙烷羧酸氧化酶 2458.66 c35274.graph_c0 叶绿体叶绿素a/b结合蛋白 2268.96 c8931.graph_c0 铁氧还蛋白 2057.79 c35272.graph_c0 叶绿体叶绿素a/b结合蛋白 2011.20 -
[1] 党选民, 曹振木, 杨(龙天), 等.热带珍稀植物辣木的特性及其开发利用[J].中国农学通报, 2004, 20(20):133-136. http://cpfd.cnki.com.cn/Article/CPFDTOTAL-ZGNX200400001033.htm [2] ANWAR F, LATIF S, ASHRAF M, et al.Moringa oleifera:a food plant with multiple medicinal uses[J]. Phytotherapy Research, 2007, 21(1):17-25. doi: 10.1002/(ISSN)1099-1573 [3] MISHRA S, SINGH P, SINGH S. Nutritional and medicinal value of Moringa oleifera leaves:potential and prospects[J]. Forestry Bulletin, 2011, 11(1):46-58. http://downloads.hindawi.com/journals/jt/2014/786979.xml [4] SUJATHA B K, POONAM P. Moringa Oleifera-Nature's Gold[J]. Imperial Journal of Interdisciplinary Research, 2017, 3(5):1175-1179. doi: 10.1556/1646.8.2016.3.7 [5] MOYO B, MASIKA P, HUGO A, et al. Nutritional characterization of Moringa (Moringa oleifera Lam.) leaves[J]. African Journal of Biotechnology, 2011, 10(60):12925-12933. doi: 10.5897/AJB [6] 刘凤霞, 王苗苗, 赵有为, 等.辣木中功能性成分提取及产品开发的研究进展[J].食品科学, 2015, 36(19):282-286. doi: 10.7506/spkx1002-6630-201519051 [7] MISHRA S P, SINGH P, SINGH S. Processing of Moringa oleifera leaves for human consumption[J]. Bulletin of Environment, Pharmacology and Life Sciences, 2012, 2(1):28-31. http://downloads.hindawi.com/journals/jt/2014/786979.xml [8] FAHEY J W. Moringa oleifera:a review of the medical evidence for its nutritional, therapeutic, and prophylactic properties. part 1[J]. Trees for life Journal, 2005, (1):5. doi: 10.1128/jmbe.v18i3.1306 [9] 彭磊, 田洋, 解静, 等.世界辣木发展现状及市场前景分析[J].世界农业, 2015, (9):143-146. http://d.wanfangdata.com.cn/Periodical_shijny201505027.aspx [10] WATERMAN C, CHENG D M, ROJAS-SILVA P, et al. Stable, water extractable isothiocyanates from Moringa oleifera leaves attenuate inflammation in vitro[J]. Phytochemistry, 2014, 103(Supplement C):114-122. https://es.scribd.com/document/119809815/JMPR-15-August-2012-Issue [11] JUNG I L. Soluble extract from Moringa oleifera leaves with a new anticancer activity[J]. PLoS ONE, 2014, 9(4):e95492. doi: 10.1371/journal.pone.0095492 [12] RICHTER N, SIDDHURAJU P, BECKER K. Evaluation of nutritional quality of moringa (Moringa oleifera Lam.) leaves as an alternative protein source for Nile tilapia (Oreochromis niloticus L.)[J]. Aquaculture, 2003, 217(1):599-611. http://www.ingentaconnect.com/content/els/00448486/2003/00000217/00000001/art00497 [13] 刘子记, 孙继华, 刘昭华, 等.特色植物辣木的应用价值及发展前景分析[J].热带作物学报, 2014, 35(9):1871-1878. http://www.cnki.com.cn/Article/CJFDTotal-RDNK201506003.htm [14] 吴崸, 蔡志华, 魏烨昕, 等.辣木作为新型植物性蛋白质饲料的研究进展[J].动物营养学报, 2013, 25(3):503-511. http://www.docin.com/p-747955573.html [15] TIAN Y, ZENG Y, ZHANG J, et al. High quality reference genome of drumstick tree (Moringa oleifera Lam.), a potential perennial crop[J]. Science China Life Sciences, 2015, 58(7):627-638. doi: 10.1007/s11427-015-4872-x [16] 田洋, 董扬, 盛军, 等.辣木转录因子家族初步鉴定与分析[J].云南农业大学学报:自然科学版, 2016, 31(6):450-461. http://www.cqvip.com/QK/94499X/201603/668956714.html [17] KONDO T, FUJIKAWA Y, UEDA A, et al. Cloning and gene expression analysis of ascorbic acid biosynthesis enzymes in Moringa oleifera[J]. African Journal of Agricultural Research, 2015, 10(22):2274-2285. doi: 10.5897/AJAR [18] DENG L T, WU Y L, LI J C, et al. Screening reliable reference genes for RT-qPCR analysis of gene expression in Moringa oleifera[J]. PLoS ONE, 2016, 11(8):e0159458. doi: 10.1371/journal.pone.0159458 [19] 邓丽婷. 基于转录组测序的辣木内参基因筛选和三个代谢途径相关基因的表达分析[D]. 广州: 华南农业大学, 2016: 48-49. http://cdmd.cnki.com.cn/Article/CDMD-10564-1016923469.htm [20] FANG Z Z, ZHOU D R, YE X F, et al. Identification of candidate anthocyanin-related genes by transcriptomic analysis of 'Furongli' plum (Prunus salicina Lindl.) during fruit ripening using RNA-Seq[J]. Frontiers in Plant Science, 2016, 7(166):1338. https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5005409/ [21] GRABHERR M G, HAAS B J, YASSOUR M, et al. Full-length transcriptome assembly from RNA-Seq data without a reference genome[J]. Nature Biotechnology, 2011, 29(7):644-652. doi: 10.1038/nbt.1883 [22] LANGMEAD B, TRAPNELL C, POP M, et al. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome[J]. Genome Biology, 2009, 10(3):1-10. http://www.oalib.com/paper/185058 [23] LI B, DEWEY C. RSEM:accurate transcript quantification from RNA-Seq data with or without a reference genome[J]. BMC Bioinformatics, 2011, 12(1):323. doi: 10.1186/1471-2105-12-323 [24] ONESA A, GÖTZ S, GARCÍA-GÓMEZ J M, et al. Blast2GO:a universal tool for annotation, visualization and analysis in functional genomics research[J]. Bioinformatics, 2005, 21(18):3674-3676. doi: 10.1093/bioinformatics/bti610 [25] YE J, FANG L, ZHENG H, et al. WEGO:a web tool for plotting GO annotations[J]. Nucleic Acids Res, 2006, 34(S2):W293-W297. https://academic.oup.com/nar/article/34/suppl_2/W293/2505468/WEGO-a-web-tool-for-plotting-GO-annotations [26] 张少平, 邱珊莲, 郑云云, 等.紫色黄秋葵转录组功能基因测序及分析[J].核农学报, 2017, 31(4):643-653. doi: 10.11869/j.issn.100-8551.2017.04.0643 [27] 向君亮, 黄殷.薏苡幼苗叶片转录组分析[J].农业生物技术学报, 2017, 25(3):386-396. http://www.jabiotech.org/CN/abstract/abstract10663.shtml [28] 武红霞, 许文天, 罗纯, 等.芒果果实转录组数据组装及基因功能注释简[J].热带作物学报, 2016, 37(11):2191-2198. doi: 10.3969/j.issn.1000-2561.2016.11.025 [29] 蔡年辉, 邓丽丽, 许玉兰, 等.基于高通量测序的云南松转录组分析[J].植物研究, 2016, 36(1):75-83. doi: 10.7525/j.issn.1673-5102.2016.01.011 [30] 姜福星, 杨丽娟, 高素萍, 等.碰碰香叶片的转录组测序分析[J].分子植物育种, 2015, 13(10):2310-2319. http://kns.cnki.net/KCMS/detail/detail.aspx?filename=fzzw201510025&dbname=CJFD&dbcode=CJFQ [31] 吴超, 彭娟, 向林, 等.基于高通量测序的铁皮石斛叶片转录组分析[J].分子植物育种, 2012, 14(12):3334-3346. http://kns.cnki.net/KCMS/detail/detail.aspx?filename=fzzw201612009&dbname=CJFD&dbcode=CJFQ [32] 王兴春, 谭河林, 陈钊, 等.基于RNA-Seq技术的连翘转录组组装与分析及SSR分子标记的开发[J].中国科学:生命科学, 2015, 45(3):301-310. http://kns.cnki.net/KCMS/detail/detail.aspx?filename=jcxk201503008&dbname=CJFD&dbcode=CJFQ -






 下载:
下载: