Codon Bias of ω-3 Fatty Acid Desaturase Gene (FAD7) in Musa itinerans
-
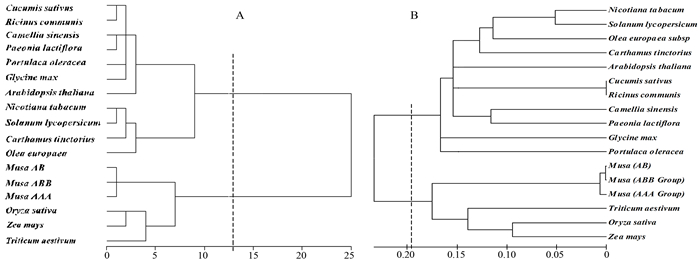
摘要: 为了解FAD7的密码子使用特性,试验采用CodonW、SPSS19.0、MEGA5.0等软件和EMBOSS在线程序对野生蕉FAD7密码子偏性进行分析,同时与其他单/双子叶植物FAD7和模式生物基因组进行比较。结果显示,野生蕉FAD7密码子偏性水平较弱,密码子组成和结尾偏好使用G或C,而CUC、CAG和AGG等在该基因中具有较高的使用频率;物种间FAD7比较结果发现,单子叶植物FAD7密码子组成明显偏好G或C,而双子叶植物则完全相反;基于密码子使用频率FAD7聚类结果与CDS分类结果一致,均能将单/双子叶植物区分开来;此外,大肠杆菌原核表达受体系统可作为野生蕉FAD7基因异源表达理想试验体系。研究结果为野生蕉FAD7后续结构和功能研究提供了科学依据。
-
关键词:
- 野生蕉 /
- 密码子偏性 /
- ω-3脂肪酸去饱和酶 /
- 聚类分析
Abstract: The codon bias of ω-3 fatty acid desaturase gene, FAD7, of Musa itinerans was analyzed using CodonW, SPSS19.0, MEGA5.0, and the EMBOSS online program. It was also compared with the FAD7s from different plants and genomes of model organisms. The results showed the codon bias of FAD7 in M. itinerans to be low and leaning toward the synonymous codons with G or C. And, it appeared that the codons CUC, CAG and AGG received a high usage frequency in the gene. In comparison with the FAD7s of different plants, the components of its monocotyledons were bias toward the use G or C, while dicotyledons the opposite. A cluster analysis on the FAD7s based on codon usage bias and CDS showed a same result. Hence, the dicotyledons and monocotyledons in the gene could be satisfactorily differentiated. Based on the frequency of codon usage, the prokaryotic expression system seemed to be suitable for heterologous expression of the FAD7 in M. itinerans.-
Key words:
- Musa itinerans /
- codon bias /
- ω-3 fatty acid desaturase /
- cluster analysis
-
表 1 野生蕉FAD7同义密码子相对使用度
Table 1. RSCU of FAD7 in M. itinerans

表 2 物种间FAD7密码子偏性参数
Table 2. Codon bias of FAD7s in various plants

表 3 野生蕉FAD7和模式生物基因组密码子偏性比较
Table 3. Comparison of codon biases in FAD7s of M. itinerans and genomes of model organisms

-
[1] BULMER M. The selection-mutation-drift theory of synonymous codon usage[J]. Genetics, 1991, 129(3):897-907. https://www.ncbi.nlm.nih.gov/pubmed/1752426?access_num=1752426&link_type=MED [2] LI W H. Models of nearly neutral mutations with particular implications for nonrandom usage of synonymous codons[J]. Journal of molecular evolution, 1987, 24(24):337-345. doi: 10.1007/BF02134132 [3] KIMURA M. Possibility of extensive neutral evolution under stabilizing selection with special reference to nonrandom usage of synonymous codons[J]. Proceedings of the National Academy of Sciences, 1981, 78(9):5773-5777. doi: 10.1073/pnas.78.9.5773 [4] 王安邦, 金志强, 刘菊华, 等.香蕉寒害研究现状及展望[J].生物技术通报, 2014, (8):28-33. http://www.cnki.com.cn/Article/CJFDTOTAL-SWJT201408005.htm [5] 赖钟雄, 陈源, 林玉玲, 等.三明野生蕉基本生物学特性调查[J].亚热带农业研究, 2006, 2(4):241-244. http://www.cnki.com.cn/Article/CJFDTOTAL-GZZZ200604000.htm [6] 赖志宸, 赖恭梯, 张群林, 等.野生蕉(Musa spp., AB group)抗寒基因FAD7的分子克隆与功能分析[J].热带作物学报, 2013, 34(10):1947-1954. doi: 10.3969/j.issn.1000-2561.2013.10.016 [7] 赖恭梯. 福建香蕉种质资源试管保存及野生蕉ISSR与抗寒性分析[D]. 福州: 福建农林大学, 2014. [8] 时慧, 王玉, 杨路成, 等.茶树抗寒调控转录因子ICE1密码子偏性分析[J].园艺学报, 2012, 39(7):1341-1352. http://www.cnki.com.cn/Article/CJFDTOTAL-YYXB201207017.htm [9] 郭秀丽, 王玉, 杨路成, 等.茶树CBF1基因密码子使用特性分析[J].遗传, 2012, 34(12):1614-1623. http://www.cnki.com.cn/Article/CJFDTOTAL-YCZZ201212020.htm [10] MCINERNEY J O. Replicational and transcriptional selection on codon usage in Borrelia burgdorferi[J]. Proceedings of the National Academy of Sciences of the United States of America, 1998, 95(18):10698-10703. doi: 10.1073/pnas.95.18.10698 [11] WONG G K, WANG J, TAO L, et al. Compositional gradients in Gramineae genes[J]. Genome research, 2002, 12(6):851-856. doi: 10.1101/gr.189102 [12] CHIAPELLO H, LISACEK F, CABOCHE M, et al. Codon usage and gene function are related in sequences of Arabidopsis thaliana[J]. Gene, 1998, 209(1-2):655-668. https://www.researchgate.net/profile/Alain_Henaut/publication/13700036_Codon_usage_and_gene_function_are_related_in_sequences_of_Arabidopsis_thaliana/links/0fcfd5006d1c72af11000000.pdf [13] MURRAY E E, LOTZER J, EBERLE M. Codon usage in plant genes[J]. Nucleic acids research, 1989, 17(2):477-498. doi: 10.1093/nar/17.2.477 [14] NOVEMBRE J A. Accounting for background nucleotide composition when measuring codon usage bias[J]. Molecular Biology & Evolution, 2002, 19(8):1390-1394. http://www.oalib.com/references/12467229 [15] 赖瑞联, 林玉玲, 钟春水, 等.龙眼生长素受体基因TIR1密码子偏好性分析[J].园艺学报, 2016, 43(4):771-780. http://www.cnki.com.cn/Article/CJFDTOTAL-YYXB201604019.htm [16] ZHOU H, WANG H, HUANG L F, et al. Heterogeneity in codon usages of sobemovirus genes[J]. Archives of virology, 2005, 150(8):1591-1605. doi: 10.1007/s00705-005-0510-4 [17] 聂江婷, 白云凤, 贺飞燕, 等.籽粒苋丙酮酸磷酸二激酶(PPDK)基因的密码子偏好性[J].植物学报, 2014, 49(6):672-681. http://www.cnki.com.cn/Article/CJFDTOTAL-ZWXT201406004.htm [18] 李平, 白云风, 冯瑞云, 等.籽粒苋苹果酸酶(NAD-ME)基因密码子偏好性分析[J].应用与环境生物学报, 2011, 17(1):12-17. http://www.cnki.com.cn/Article/CJFDTOTAL-YYHS201101004.htm -






 下载:
下载:

