Codon Usage Bias of Chloroplast Genome in Liparis bootanensis
-
摘要:
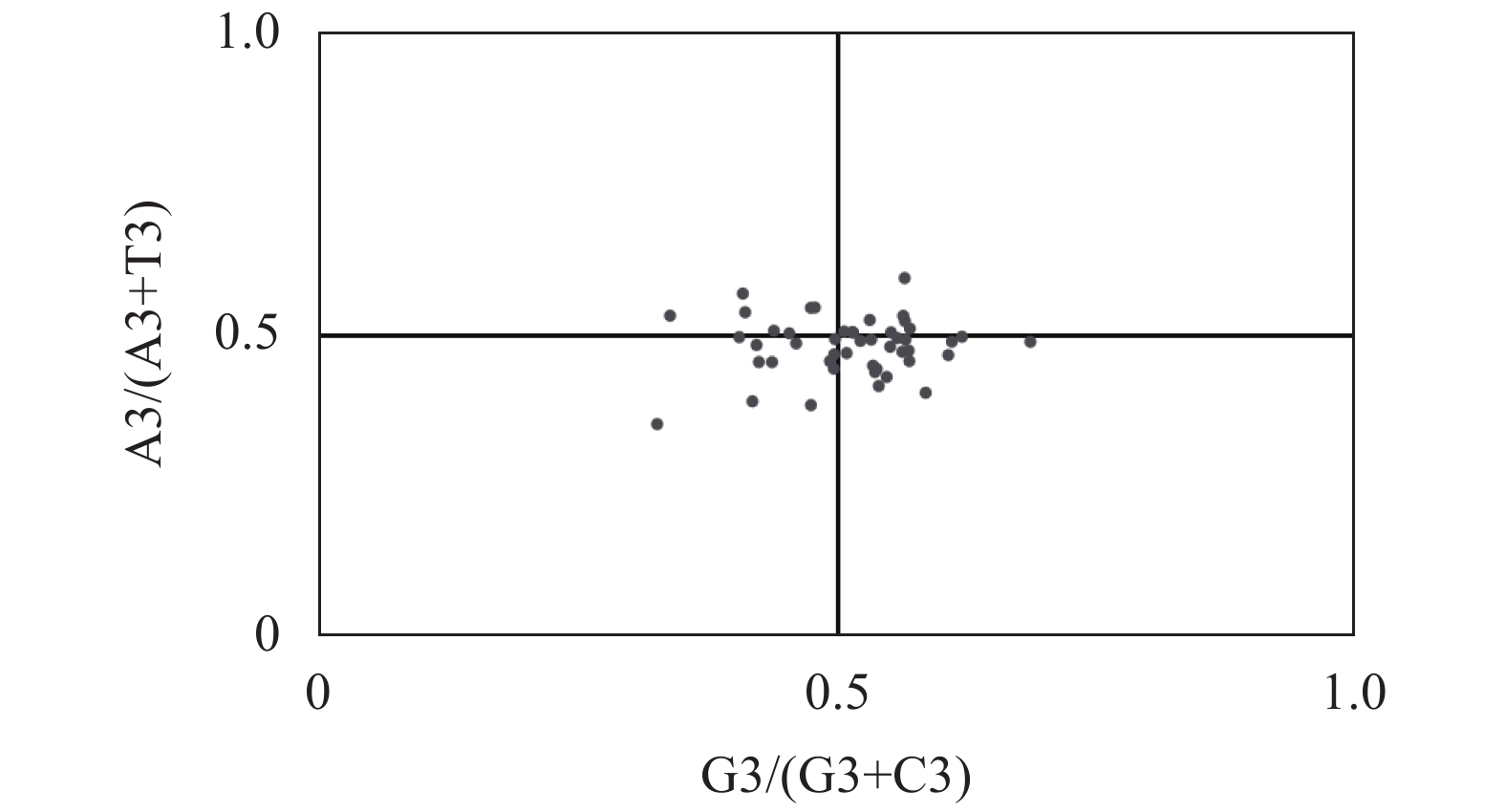
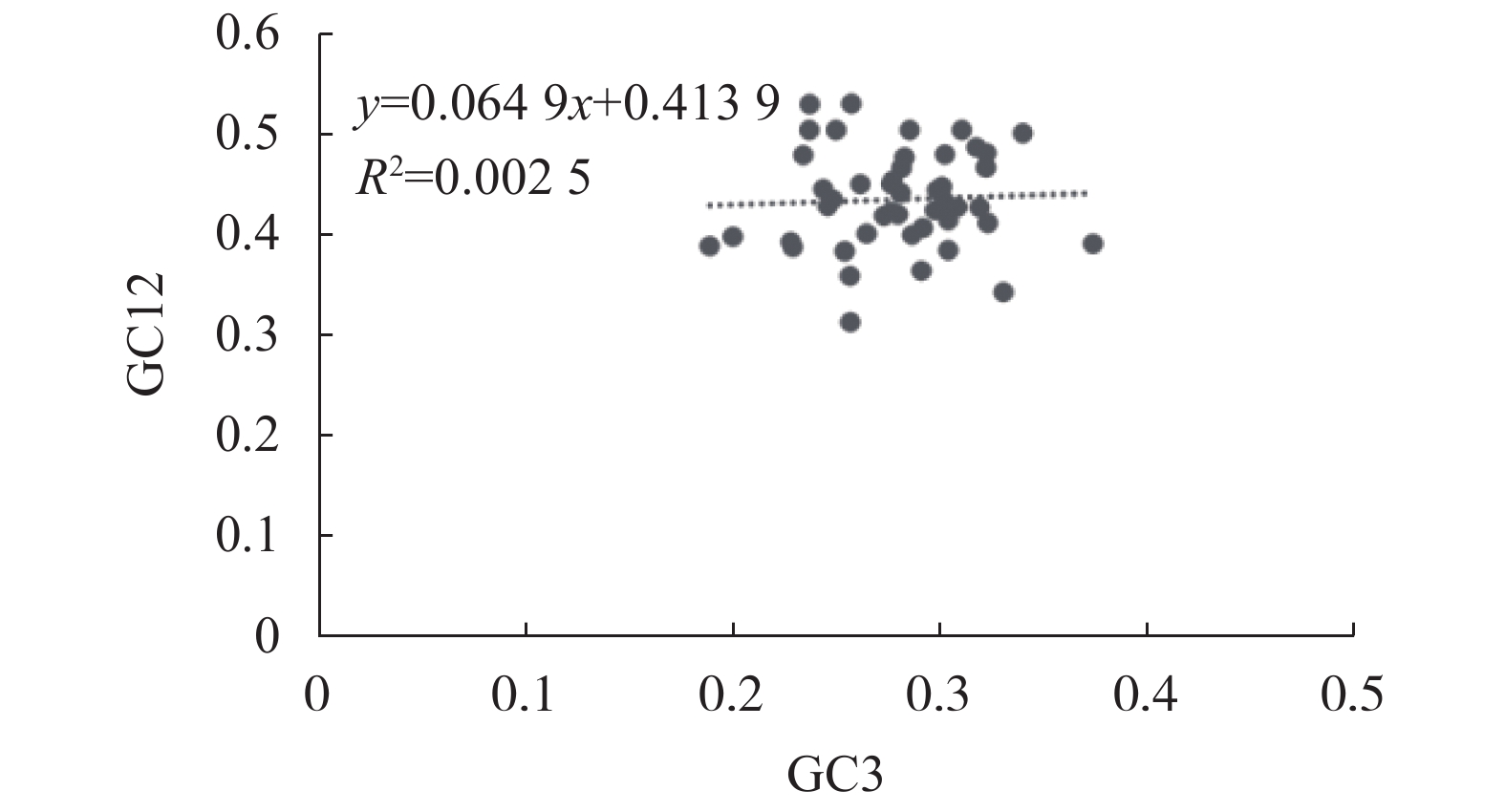
目的 分析镰翅羊耳蒜叶绿体基因组密码子偏好性,并探讨影响密码子偏好性形成的主要因素。 方法 从镰翅羊耳蒜叶绿体基因组中筛选出47条编码序列,通过CodonW和CUSP软件计算不同基因的GC含量,分析密码子使用模式。 结果 GC3含量为27.99%,低于GC1(47.00%)和GC2(39.41%);ENC取值范围为40.85~56.80,平均值为47.27;ENC与GC1、GC2之间均无显著相关,与GC3呈极显著正相关;GC12与GC3的相关性不显著,相关系数为0.05;47%基因的ENC比值频数分布在−0.05~0.05区间,53%基因的ENC比值频数分布在−0.05~0.05区间之外;碱基的使用频率为A>T、G>C;17个密码子被确定为最优密码子。 结论 镰翅羊耳蒜叶绿体基因组密码子对A或T碱基结尾有偏好性;密码子偏好性较弱;密码子偏好性主要受选择和突变作用共同影响,同时还受其他因素影响,是多因素综合影响的结果。 Abstract:Objective The coden usage bias and its infulence factor in the chloroplast genome of Liparis bootanensis were analyzed in this study. Method CodonW and CUSP software were used to analyze the codon usage bias based on 47 codon DNA sequences of the chloroplast genome of L. bootanensis. Result The GC content in the 3rd position of the codons was 27.99%, which was lower than those in the 1st and the 2nd. The effective number of codons ranged from 40.85 to 56.80, averaging 47.27. The ENC showed no significant correlation to GC1 and GC2 but a significant positive correlation to GC3. The correlation between GC12 and GC3 was not significant with a coefficient of 0.05. The frequency of ENC ratio of 47% genes was distributed between −0.05 and 0.05, while that of 53% genes outside -0.05 and 0.05 range. A PR2-plot analysis indicated the base usage frequencies to be A>T and G>C. There were 17 codons selected after screening. Conclusion The 3rd position of codons was rich in A or T. The codon usage bias was weak, complex, and affected by mutation and selection besides other factors. -
Key words:
- Liparis bootanensis /
- chloroplast genome /
- codon usage bias
-
表 1 编码基因密码子不同位置GC含量
Table 1. GC content in various positions of codon in chloroplast gene
基因 Gene GC1/% GC2/% GC3/% GCall/% ENC 基因 Gene GC1/% GC2/% GC3/% GCall/% ENC rps12 52.42 47.58 25.00 41.67 43.84 petA 53.89 35.51 26.17 38.53 48.43 psbA 49.44 43.22 32.20 41.62 43.05 rps18 37.25 45.1 30.39 37.58 43.24 atpA 54.72 40.35 23.43 39.50 43.05 rpl20 36.67 44.17 29.17 36.67 54.03 atpF 45.95 33.51 28.65 36.04 48.25 clpP 57.56 37.07 28.29 40.98 56.80 atpI 48.39 36.69 24.60 36.56 45.27 psbB 53.83 46.17 31.04 43.68 48.68 rps2 43.22 40.25 27.97 37.15 47.67 petB 47.69 41.2 30.09 39.66 43.21 rpoC2 45.91 37.29 27.30 36.83 48.68 petD 50.61 37.8 24.39 37.60 43.52 rpoC1 50.15 38.09 29.85 39.36 49.05 rpoA 45.00 34.71 26.47 35.39 48.64 rpoB 49.30 38.38 28.10 38.59 48.53 rps11 53.24 51.8 23.74 42.93 43.04 psbD 52.26 42.94 30.23 41.81 42.10 rps8 40.15 37.12 18.94 32.07 40.85 psbC 53.38 45.99 33.97 44.44 45.89 rpl14 53.66 35.77 27.64 39.02 43.44 rps14 43.56 46.53 27.72 39.27 41.37 rpl16 52.21 52.94 25.74 43.63 41.93 psaA 52.06 43.41 32.22 42.57 49.71 rps3 44.29 33.79 22.83 33.64 45.82 ycf3 47.34 39.05 24.85 37.08 53.43 rpl22 41.40 35.67 22.93 33.33 44.99 rps4 48.02 38.12 30.20 38.78 51.44 ycf2 42.53 35.21 37.34 38.36 53.23 ndhJ 47.17 37.74 30.82 38.57 53.38 ndhB 41.88 39.92 32.29 38.03 47.85 ndhK 41.53 42.80 30.08 38.14 50.43 rps7 53.85 46.15 23.72 41.24 47.64 ndhC 49.59 34.71 29.75 38.02 51.42 ccsA 34.37 38.08 29.10 33.85 47.69 atpE 50.37 42.22 28.15 40.25 44.72 ndhE 43.14 33.33 30.39 35.62 54.50 atpB 55.29 41.32 31.74 42.78 51.77 ndhG 40.11 36.16 25.42 33.9 45.14 rbcL 57.69 42.31 28.54 42.85 47.79 ndhA 42.03 37.09 20.05 33.06 42.54 accD 36.44 34.99 25.67 32.37 44.91 ndhH 48.22 35.79 27.66 37.23 47.50 ycf4 43.78 41.08 31.89 38.92 48.68 ycf1 35.72 26.67 25.69 29.36 46.69 cemA 41.74 26.52 33.04 33.77 47.70 平均值 AVG 47.00 39.41 27.99 38.13 47.27 表 2 编码基因密码子参数的相关性
Table 2. Correlations among individual related parameters of genes
项目 Items GC1 GC2 GC3 GCall ENC N GC1 1.000 GC2 0.390** 1.000 GC3 0.047 0.035 1.000 GCall 0.802** 0.758** 0.396** 1.000 ENC 0.022 −0.277 0.455** 0.029 N −0.126 −0.286 0.296* −0.118 0.196 1.000 注:*表示在0.05水平上显著相关;**表示在0.01水平上极显著相关。
Note: * means significant correlation at 0.05 level; ** means extremely significant correlation at 0.01 level.表 3 相对同义密码子使用度分析
Table 3. RSCU analysis on protein coding region
氨基酸Amino acid 密码子 Coden 数量 Number RSCU 氨基酸 Amino acid 密码子 Coden 数量 Number RSCU Phe UUU 649 1.37 Ser UCU 410 1.71 UUC 359 0.63 UCC 231 0.98 Leu UUA 584 2.09 UCA 283 1.25 UUG 379 1.16 UCG 114 0.43 CUU 384 1.31 Pro CCU 287 1.47 CUC 127 0.38 CCC 172 0.89 CUA 259 0.74 CCA 206 1.15 CUG 126 0.32 CCG 75 0.31 AUU 770 1.48 Thr ACU 379 1.69 Ile AUC 312 0.57 ACC 154 0.68 AUA 492 0.95 ACA 289 1.16 Met AUG 432 1.00 ACG 103 0.47 Val GUU 369 1.45 Ala GCU 444 1.82 GUC 119 0.39 GCC 133 0.59 GUA 377 1.44 GCA 309 1.23 GUG 165 0.73 GCG 93 0.36 Tyr UAU 527 1.68 Cys UGU 157 1.51 UAC 130 0.32 UGC 53 0.36 Ter UAA 22 1.40 Ter UGA 11 0.70 UAG 14 0.89 Trp UGG 322 0.87 His CAU 330 1.45 Arg CGU 260 1.60 CAC 97 0.47 CGC 63 0.37 Gln CAA 528 1.59 CGA 246 1.31 CAG 162 0.41 CGG 83 0.43 Asn AAU 695 1.50 AGA 359 1.71 AAC 190 0.45 AGG 116 0.59 Lys AAA 772 1.50 Ser AGU 292 1.31 AAG 249 0.41 AGC 75 0.31 Asp GAU 638 1.62 Gly GGU 405 1.30 GAC 131 0.38 GGC 122 0.41 Glu GAA 822 1.52 GGA 499 1.68 GAG 271 0.48 GGG 206 0.62 注:Phe:苯丙氨酸,Leu:亮氨酸,Ile:异亮氨酸,Met:蛋氨酸, Val:缬氨酸,Tyr:酪氨酸,His:组氨酸,Gln:谷氨酰胺, Asn:天冬酰胺,Lys:赖氨酸,Asp:天冬氨酸,Glu:谷氨酸,Ser:丝氨酸, Pro:脯氨酸,Thr:苏氨酸,Ala:丙氨酸,Cys:半胱氨酸,Trp:色氨酸,Arg:精氨酸,Gly:甘氨酸,Ter:终止密码子。表5同。
Note: Phe: phenylalanine; Leu: leucine; Ile: isoleucine; Met: methionine; Val: valine; Tyr: tyrosine; His: histidine; Gln: glutamine; Asn: asparagine; Lys: lysine; Asp: aspartic acid; Glu: glutamic acid; Ser: serine; Pro: proline; Thr: threonine; Ala: alanine; Cys: cysteine; Trp: tryptophane; Arg: argnine; Gly: glycine; Ter: termination codon. Same for Table 5.表 4 ENC比值频数分布
Table 4. Distribution of ENC ratios
组限
Class limits组中值
Class mid value组数
Frequency number组频
Frequency−0.25~−0.15 −0.20 1 0.02 −0.15~−0.05 −0.75 5 0.11 −0.05~0.05 0.00 22 0.47 0.05~0.15 0.10 19 0.40 合计 total 47 1.00 表 5 最优密码子分析
Table 5. Analysis on preferred codon
氨基酸
Amino acid密码子
Coden高表达基因
High expressed gene低表达基因
Low expressed geneΔRSCU 氨基酸
Amino acid密码子
Coden高表达基因
High expressed gene低表达基因
Low expressed geneΔRSCU 数量
NumberRSCU 数量
NumberRSCU 数量
NumberRSCU 数量
NumberRSCU Phe UUU* 53 1.61 25 1.46 0.15 Ser UCU*** 28 1.86 11 1.15 0.71 UUC 19 0.39 9 0.54 -0.15 UCC* 12 1.14 9 0.93 0.21 Leu UUA 47 1.70 21 1.89 -0.19 UCA 16 1.07 14 1.55 −0.48 UUG* 17 1.05 9 0.81 0.24 UCG 2 0.28 5 0.57 −0.29 CUU* 26 1.76 18 1.48 0.28 Pro CCU*** 21 1.89 10 0.91 0.98 CUC 6 0.43 5 0.37 0.06 CCC*** 10 1.03 3 0.29 0.74 CUA 24 0.84 10 0.90 −0.06 CCA 11 0.93 9 0.90 0.03 CUG 8 0.22 7 0.56 −0.34 CCG 2 0.15 3 0.29 −0.14 Ile AUU* 47 1.36 28 1.22 0.14 Thr ACU* 23 1.53 13 1.36 0.17 AUC 10 0.30 13 0.61 −0.31 ACC* 11 0.97 8 0.76 0.21 AUA* 39 1.33 26 1.16 0.17 ACA* 18 1.37 9 1.14 0.23 Met AUG 27 1.00 20 1.00 0.00 ACG 3 0.13 6 0.74 −0.61 Val GUU*** 27 2.19 17 1.55 0.64 Ala GCU 37 1.69 27 2.08 −0.39 GUC* 10 0.41 4 0.29 0.12 GCC 6 0.47 8 0.52 −0.05 GUA 20 1.15 14 1.43 −0.28 GCA*** 26 1.50 14 0.93 0.57 GUG 4 0.24 5 0.73 −0.49 GCG 6 0.34 6 0.47 −0.13 Tyr UAU* 31 1.88 30 1.67 0.21 Cys UGU 10 1.73 5 1.73 0.00 UAC 4 0.12 6 0.33 −0.21 UGC 2 0.27 2 0.27 0.00 Ter UAA 3 1.80 3 1.80 0.00 Ter UGA 1 0.60 1 0.60 0.00 UAG 1 0.60 1 0.60 0.00 Trp UGG 28 1.00 13 0.80 0.20 His CAU 15 1.50 15 1.54 −0.04 Arg CGU** 21 1.60 12 1.21 0.39 CAC 2 0.50 5 0.46 0.04 CGC 3 0.26 3 0.36 −0.10 Gln CAA 28 1.61 24 1.75 −0.14 CGA* 21 1.64 13 1.38 0.26 CAG* 7 0.39 4 0.25 0.14 CGG 1 0.08 7 0.79 −0.71 Asn AAU 27 1.50 33 1.82 −0.32 AGA** 22 1.83 15 1.50 0.33 AAC** 10 0.50 4 0.18 0.32 AGG 7 0.60 8 0.76 −0.16 Lys AAA 34 1.61 23 1.61 0.00 Ser AGU** 17 1.37 12 1.21 0.16 AAG 10 0.39 7 0.39 0.00 AGC 2 0.28 5 0.59 −0.16 Asp GAU 18 0.97 31 1.92 −0.95 Gly GGU*** 35 1.31 7 0.68 0.63 GAC* 8 0.24 2 0.08 0.16 GGC 13 0.44 8 0.60 −0.16 Glu GAA*** 44 1.76 32 1.03 0.73 GGA 29 1.73 18 1.92 −0.19 GAG 6 1.03 15 0.97 0.06 GGG 11 0.52 5 0.79 −0.27 注:_表示RSCU 值大于1 的密码子; *表示△RSCU≥0.08; **表示ΔRSCU≥0.3; ***表示ΔRSCU≥0.5。
Note: Underlining indicates RSCU>1; * means △RSCU≥0.08; ** means △RSCU≥0.3; *** means ΔRSCU≥0.5. -
[1] CHEN S C, WOOD J J, ORMEROD P. Liparis L. C. Richard. In: WU Z Y, RAVEN P H, HONG D. (Eds.) Flora of China, vol. 25[M]. Science Press, Beijing & Missouri Botanical Garden Press, St. Louis, 2009: 211–228. [2] 乔永刚, 贺嘉欣, 王勇飞, 等. 药用植物苦参的叶绿体基因组及其特征分析 [J]. 药学学报, 2019, 54(11):2106−2112.QIAO Y G, HE J X, WANG Y F, et al. Analysis of chloroplast genome and its characteristics of medicinal plant Sophora flavescens [J]. Acta Pharmaceutica Sinica, 2019, 54(11): 2106−2112.(in Chinese) [3] DANIELL H, KHAN M S, ALLISON L. Milestones in chloroplast genetic engineering: an environmentally friendly era in biotechnology [J]. Trends in Plant Science, 2002, 7(2): 84−91. doi: 10.1016/S1360-1385(01)02193-8 [4] LIANG W, GUO X, NAGLE D G, et al. Genus Liparis: A review of its traditional uses in China, phytochemistry and pharmacology [J]. Journal of Ethnopharmacology, 2019, 234: 154−171. doi: 10.1016/j.jep.2019.01.021 [5] 国家中医药管理局编委会. 中华本草[M]. 上海: 上海科学技术出版社, 1999. [6] 方志先, 廖朝林. 湖北恩施药用植物志(下册)[M]. 武汉: 湖北科学技术出版社, 2006. [7] 吴润. 镰翅羊耳蒜中化学成分的研究[D]. 成都: 西南交通大学, 2017.WU R. Study on chemical constituents of Liparis bootanensis[D]. Chengdu: Southwest Jiaotong University, 2017. (in Chinese). [8] 林爱英. 福建3种野生兰科植物繁殖生物学的初步研究[D]. 福州: 福建师范大学, 2015.LIN A Y. A preliminary study on reproductive biology of three wild orchids in Fujian province[D]. Fuzhou: Fujian Normal University, 2015. (in Chinese). [9] LIU J F. The complete chloroplast genome sequence of Liparis bootanensis (Orchidaceae) [J]. Mitochondrial DNA Part B, 2020, 5(3): 2058−2059. doi: 10.1080/23802359.2020.1763866 [10] BULMER M. The selection-mutation-drift theory of synonymous codon usage [J]. Genetics, 1991, 129: 897−907. doi: 10.1093/genetics/129.3.897 [11] ROMERO H, ZAVALA A, MUSTO H. Codon usage in Chlamydia trachomatis is the result of strand-specific mutational biases and a complex pattern of selective forces [J]. Nucleic Acids Research, 2000, 28(10): 2084−2090. doi: 10.1093/nar/28.10.2084 [12] HIRAOKA Y, KAWAMATA K, HARAGUCHI T, et al. Codon usage bias is correlated with gene expression levels in the fission yeast Schizosaccharomyces pombe [J]. Genes to Cells, 2009, 14(4): 499−509. doi: 10.1111/j.1365-2443.2009.01284.x [13] SHARP P M, EMERY L R, ZENG K. Forces that influence the evolution of Codon bias [J]. Philosophical Transactions of the Royal Society of London Series B, Biological Sciences, 2010, 365(1544): 1203−1212. doi: 10.1098/rstb.2009.0305 [14] 杨国锋, 苏昆龙, 赵怡然, 等. 蒺藜苜蓿叶绿体密码子偏好性分析 [J]. 草业学报, 2015, 24(12):171−179. doi: 10.11686/cyxb2015016YANG G F, SU K L, ZHAO Y R, et al. Analysis of Codon usage in the chloroplast genome of Medicago truncatula [J]. Acta Prataculturae Sinica, 2015, 24(12): 171−179.(in Chinese) doi: 10.11686/cyxb2015016 [15] 王婧, 王天翼, 王罗云, 等. 沙枣叶绿体全基因组序列及其使用密码子偏性分析 [J]. 西北植物学报, 2019, 39(9):1559−1572.WANG J, WANG T Y, WANG L Y, et al. Assembling and analysis of the whole chloroplast genome sequence of Elaeagnus angustifolia and its Codon usage bias [J]. Acta Botanica Boreali-Occidentalia Sinica, 2019, 39(9): 1559−1572.(in Chinese) [16] WRIGHT F. The ‘effective number of codons’ used in a gene [J]. Gene, 1990, 87(1): 23−29. doi: 10.1016/0378-1119(90)90491-9 [17] JIANG Y, DENG F, WANG H L, et al. An extensive analysis on the global Codon usage pattern of baculoviruses [J]. Archives of Virology, 2008, 153(12): 2273−2282. doi: 10.1007/s00705-008-0260-1 [18] SUEOKA N. Directional mutation pressure and neutral molecular evolution [J]. Proceedings of the National Academy of Sciences of the United States of America, 1988, 85(8): 2653−2657. doi: 10.1073/pnas.85.8.2653 [19] SUEOKA N. Near homogeneity of PR2-bias fingerprints in the human genome and their implications in phylogenetic analyses [J]. Journal of Molecular Evolution, 2001, 53(4/5): 469−476. [20] 胡莎莎, 罗洪, 吴琦, 等. 苦荞叶绿体基因组密码子偏爱性分析 [J]. 分子植物育种, 2016, 14(2):309−317.HU S S, LUO H, WU Q, et al. Analysis of Codon bias of chloroplast genome of Tartary buckwheat [J]. Molecular Plant Breeding, 2016, 14(2): 309−317.(in Chinese) [21] 续晨, 贲爱玲, 蔡晓宁. 蝴蝶兰叶绿体基因组密码子使用的相关分析 [J]. 分子植物育种, 2010, 8(5):945−950. doi: 10.3969/mpb.008.000945XU C, BEN A L, CAI X N. Analysis of synonymous Codon usage in chloroplast genome of Phalaenopsis aphrodite subsp. Formosana [J]. Molecular Plant Breeding, 2010, 8(5): 945−950.(in Chinese) doi: 10.3969/mpb.008.000945 [22] 李冬梅, 吕复兵, 朱根发, 等. 文心兰叶绿体基因组密码子使用的相关分析 [J]. 广东农业科学, 2012, 39(10):61−65. doi: 10.3969/j.issn.1004-874X.2012.10.019LI D M, LV F B, ZHU G F, et al. Analysis on Codon usage of chloroplast genome of Oncidium Gower Ramsey [J]. Guangdong Agricultural Sciences, 2012, 39(10): 61−65.(in Chinese) doi: 10.3969/j.issn.1004-874X.2012.10.019 [23] 刘慧, 王梦醒, 岳文杰, 等. 糜子叶绿体基因组密码子使用偏性的分析 [J]. 植物科学学报, 2017, 35(3):362−371. doi: 10.11913/PSJ.2095-0837.2017.30362LIU H, WANG M X, YUE W J, et al. Analysis of Codon usage in the chloroplast genome of Broomcorn millet (Panicum miliaceum L.) [J]. Plant Sclence Journal, 2017, 35(3): 362−371.(in Chinese) doi: 10.11913/PSJ.2095-0837.2017.30362 [24] INGVARSSON P K. Gene expression and protein length influence Codon usage and rates of sequence evolution in Populus tremula [J]. Molecular Biology and Evolution, 2007, 24(3): 836−844. [25] 陆奇丰, 骆文华, 黄至欢. 两种梧桐叶绿体基因组密码子使用偏性分析 [J]. 广西植物, 2020, 40(2):173−183. doi: 10.11931/guihaia.gxzw201811035LU Q F, LUO W H, HUANG Z H. Codon usage bias of chloroplast genome from two species of Firmiana Marsili [J]. Guihaia, 2020, 40(2): 173−183.(in Chinese) doi: 10.11931/guihaia.gxzw201811035 -






 下载:
下载: