Cloning and Analysis of Reverse Transcriptase Gene of Ty3-gypsy-like Retrotransposons in Sugarcane
-
摘要:
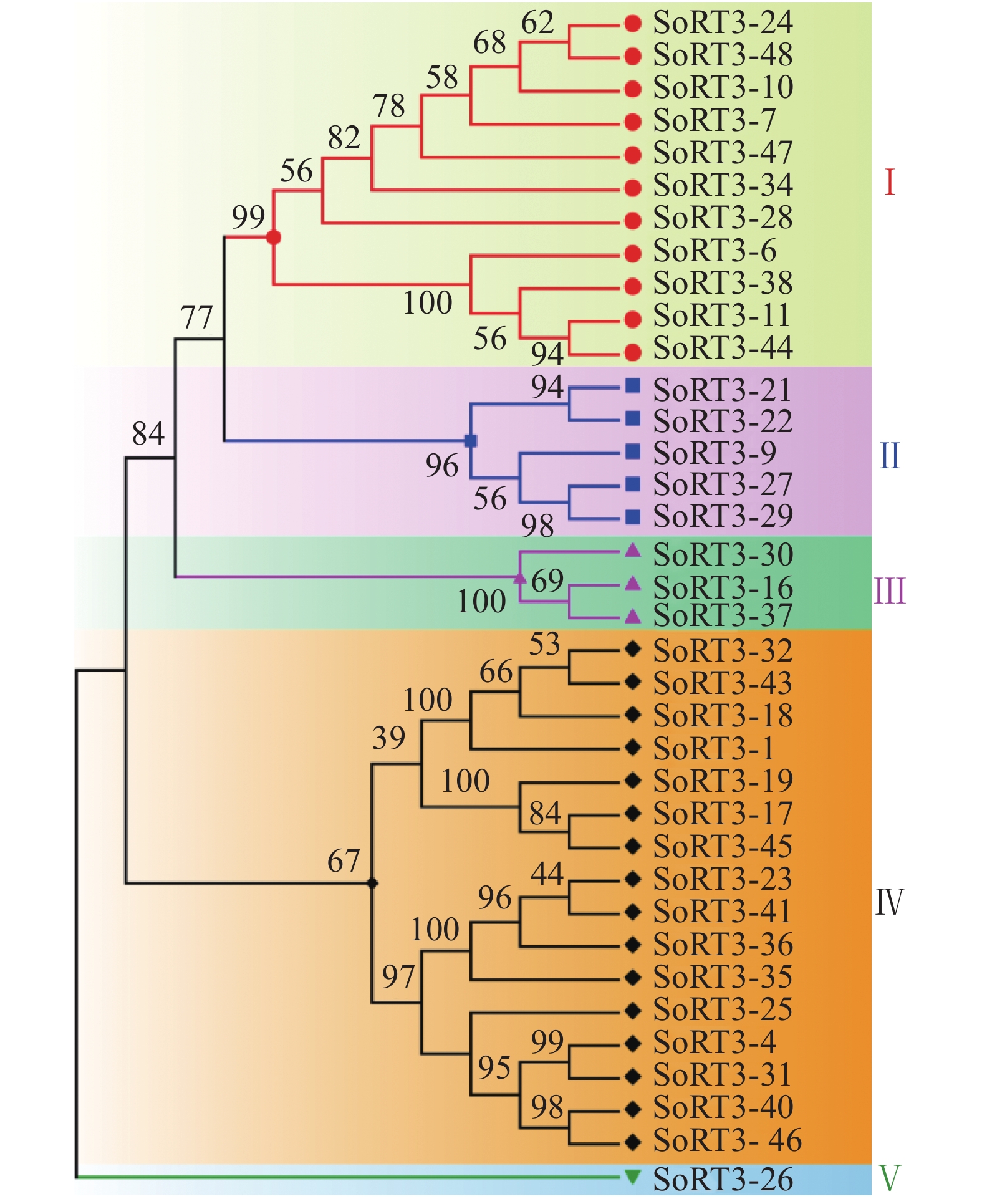
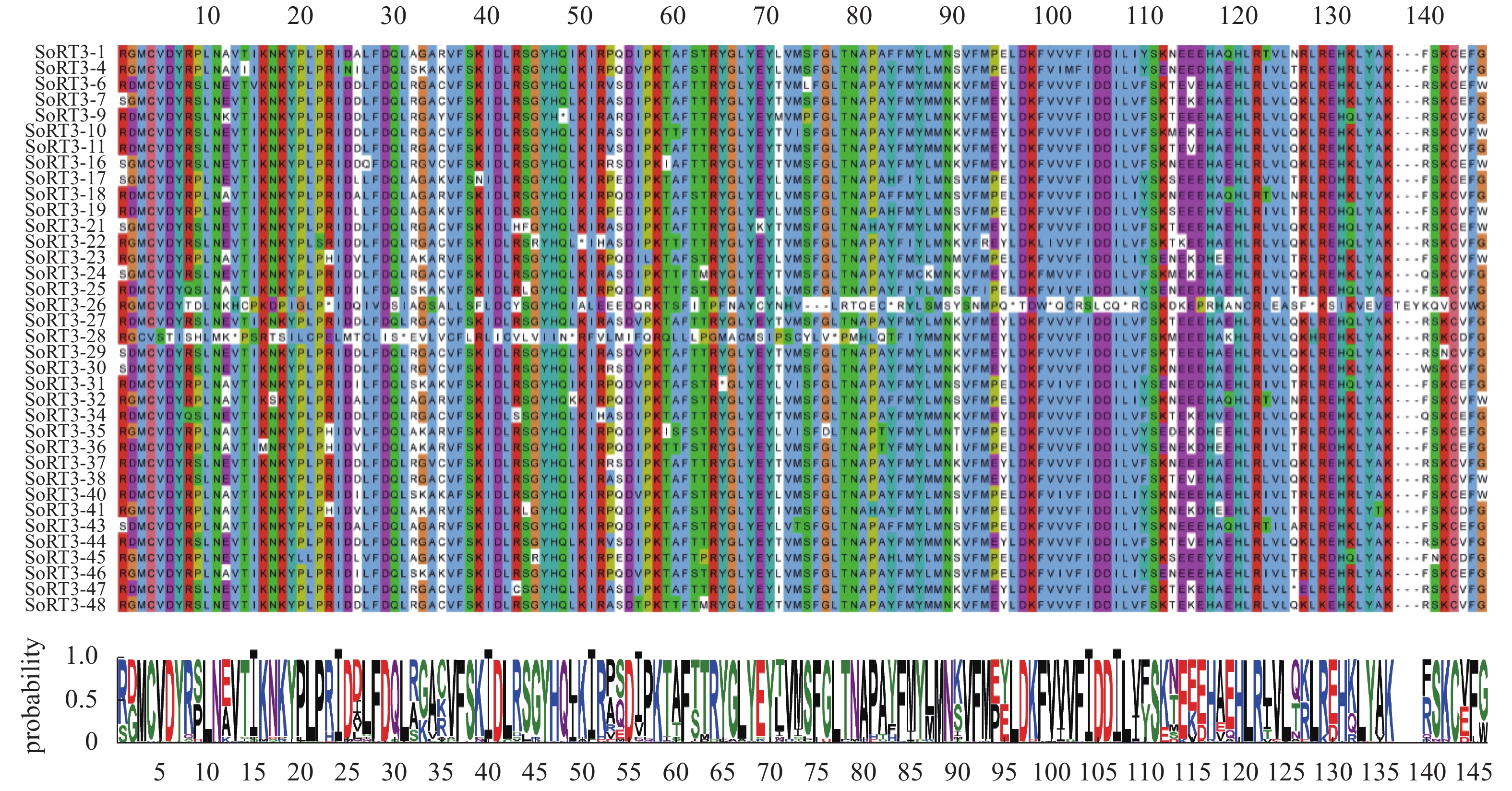
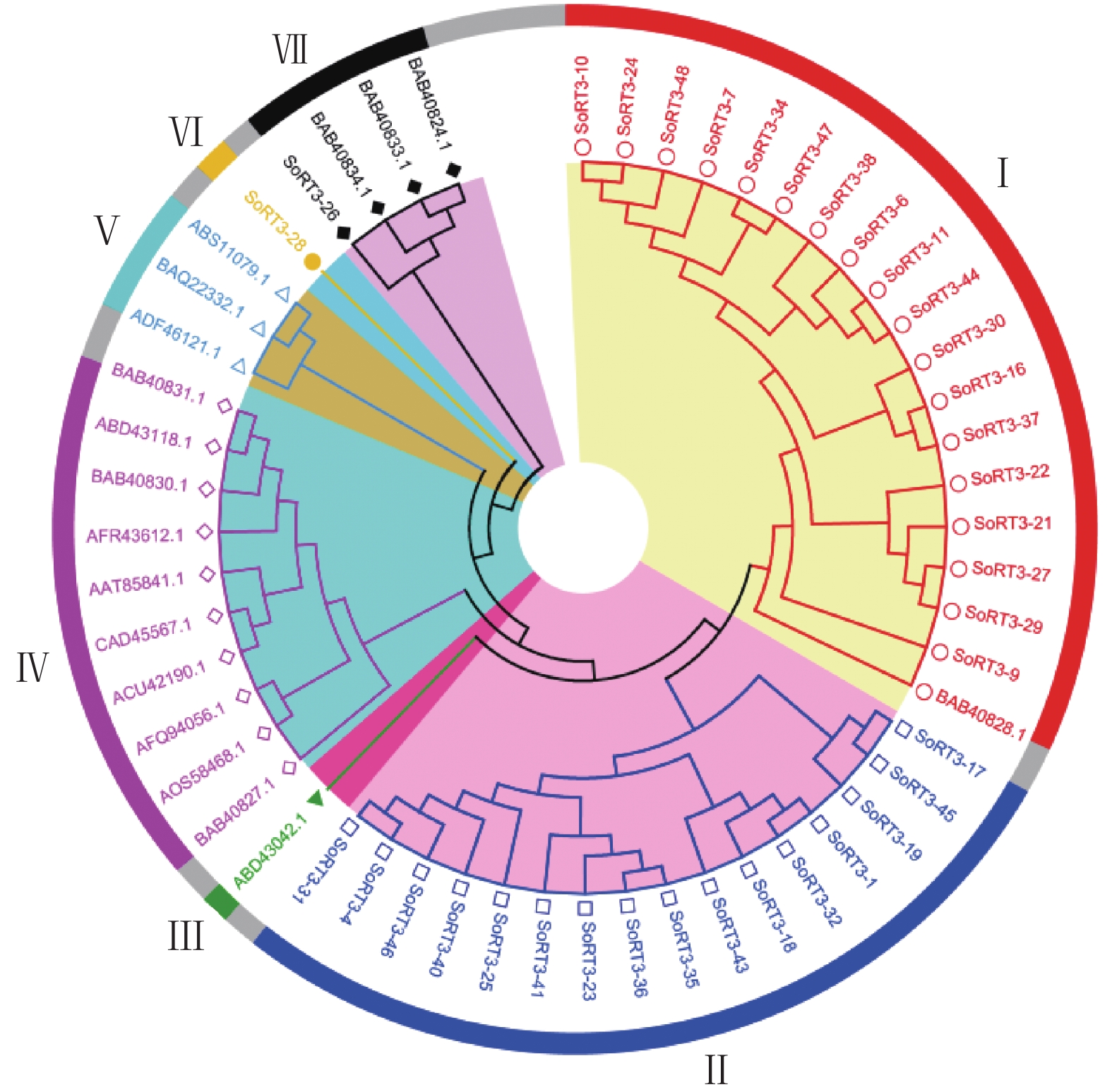
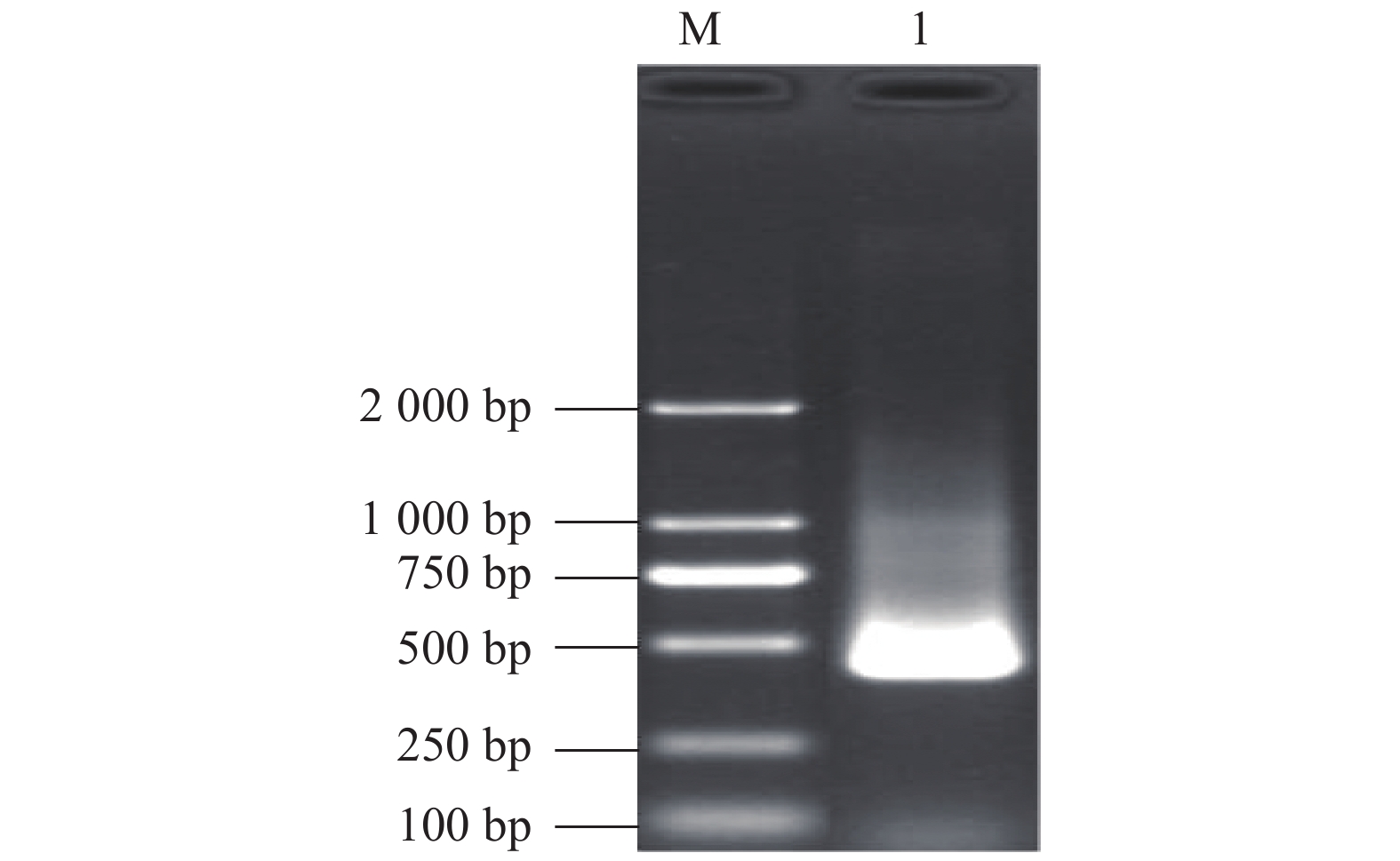
目的 从甘蔗基因组中分离Ty3-gypsy类逆转座子的RT基因序列,分析序列的特点、差异及系统进化关系,为研究其转录活性和调控功能奠定基础。 方法 根据Ty3-gypsy类逆转座子RT基因序列的保守区设计简并引物,对甘蔗品种新台糖22的基因组DNA进行PCR扩增,目的条带经回收、克隆和测序后,对所获得序列进行生物信息学分析。 结果 目的条带大小约430 bp,成功分离到36条RT基因序列,只有1条序列的长度为430 bp,其余序列的长度均为432 bp,AT所占比为56.71%~64.81%,AT与GC比值为1.31~1.84;核苷酸序列间相似性为46.2%~99.3%。聚类分析后,36条RT基因序列被划分为5个家族,家族Ⅰ和家族Ⅳ是包含序列最多的家族。翻译后,有6条序列发生了无义突变;氨基酸序列间相似性为10.1%~100%,呈现高度异质性;36条RT基因序列中的34条序列的保守基序完全一致,呈现高度保守性。各家族中代表序列的蛋白质三级结构整体构型基本类似,但在氢键数和转角数上存在着较大差别,呈现一定异质性和多态性。系统进化树显示,所有RT基因序列被分为7类,Ⅰ类中的18条甘蔗RT基因序列与拟南芥的BAB40828.1具有较高相似性,Ⅶ类中的SoRT3-26与大豆的BAB40834.1、菠菜的BAB40833.1和粳稻的BAB40824.1的亲缘关系最近,暗示在进化过程中甘蔗与这些物种植物之间可能发生了Ty3-gypsy类逆转座子的横向传递;通过比对甘蔗EST数据库,发现了10条具有转录活性的甘蔗品种新台糖22的Ty3-gypsy类逆转座子。 结论 获得甘蔗Ty3-gypsy类逆转座子RT基因序列及其具有转录活性序列,为下一步分离Ty3-gypsy类逆转座子全长序列及研究Ty3-gypsy类逆转座子转录转座活性和功能提供序列基础,也为开发甘蔗LTR逆转座子的分子标记奠定基础。 -
关键词:
- 甘蔗 /
- Ty3-gypsy类逆转座子 /
- 逆转录酶 /
- 新台糖22 /
- 异质性
Abstract:Objective The reverse transcriptase (RT) gene sequences of Ty3-gypsy-like retrotransposons were isolated from the sugarcane genome, and their characteristics, differences, and phylogenetic relationships analyzed to study the transcriptional activities and regulatory functions associated with the retrotransposons. Method Degenerate primers were designed according to the conserved region of the RT gene sequences of Ty3-gypsy-like retrotransposons. The genomic DNA of sugarcane variety, ROC22, was amplified by PCR. The recovered, cloned, and sequenced target band was analyzed by bioinformatics. Result Thirty-six RT gene sequences were successfully isolated. Only one sequence of the target bands was 430 bp in length, while the remainders were 432 bp. The proportions of AT in the sequence ranged from 56.71% to 64.81%, and the ratio of AT to GC from 1.31 to 1.84. A high heterogeneity existed among the nucleotide sequences, as the similarity was shown to be 46.2% to 99.3%. The cluster analysis divided the 36 RT gene sequences into 5 families. Family I and Family IV contained most of the sequences. After translated into amino acids, 6 sequences displayed nonsense mutations. The similarity between amino acid sequences ranged from 10.1% to 100% exhibiting a high degree of heterogeneity. The conserved motifs of 34 of the sequences were identical, indicating a highly conservative property. The overall configuration of the tertiary structure of the representative proteins in each family was basically similar, however, there were significant differences in the number of hydrogen bonds and turns, suggesting a certain degree of heterogeneity and polymorphism. The phylogenetic tree showed 7 distinct groups among the RT gene sequences. Of which, 18 in Group I were highly homology with BAB40828.1 of Arabidopsis thaliana, and SoRT3-26 in Group VII was genetically closest to the BAB40834.1 of soybean, BAB40833.1 of spinach, and BAB40824.1 of japonica rice. It indicated that a horizontal transfer of Ty3-gypsy-like retrotransposons might have occurred in evolution between sugarcane and those plant species. By comparing the sugarcane EST database, 10 Ty3-gypsy-like retrotransposons were found with transcriptional activity in ROC22. Conclusion Thirty-six RT gene sequences of Ty3-gypsy-like retrotransposons in sugarcane were obtained with 10 of them showing a transcriptional activity. The results provided the basic information for the isolation of full-length Ty3-gypsy-like retrotransposons, further understanding of the transcriptional, transposon activity and functions of the retrotransposons, and the development of sugarcane molecular markers based on LTR retrotransposons. -
Key words:
- sugarcane /
- Ty3-gypsy-like retrotransposons /
- reverse transcriptase /
- ROC22 /
- heterogeneity
-
图 4 Ty3-gypsy类逆转座子RT基因序列遗传进化树
注:树枝节点处的数值表示1 000次重复得到的自展值,树枝长度代表以邻接法估算出来的遗传距离。
Figure 4. Phylogenetic tree of RT of Ty3-gypsy-like retrotransposons
Note: data shown at branch nodes are bootstrap support from 1 000 replications. Branch lengths are proportional to genetic distances as estimated by neighbor-joining method.
表 1 Ty3-gypsy类逆转座子RT基因序列的基本信息
Table 1. Basic information on RT gene of Ty3-gypsy-like retrotransposons
序列编号
Sequence No.大小
Size/bpA+T含量
A+T content/%AT/GC 登录号
Accession No.序列编号
Sequence No.大小
Size/bpA+T含量
A+T content/%AT/GC 登录号
Accession No.SoRT3-1 432 56.71 1.31 MK830281 SoRT3-29 432 61.34 1.60 MK830300 SoRT3-4 432 60.88 1.56 MK830282 SoRT3-30 432 60.19 1.51 MK830301 SoRT3-6 432 63.89 1.77 MK830283 SoRT3-31 432 60.19 1.51 MK830302 SoRT3-7 432 60.88 1.56 MK830284 SoRT3-32 432 56.94 1.32 MK830303 SoRT3-9 432 61.34 1.59 MK830285 SoRT3-34 432 62.73 1.68 MK830304 SoRT3-10 432 63.43 1.73 MK830286 SoRT3-35 432 62.50 1.67 MK830305 SoRT3-11 432 62.04 1.63 MK830287 SoRT3-36 432 62.73 1.68 MK830306 SoRT3-16 432 60.42 1.53 MK830288 SoRT3-37 432 61.11 1.57 MK830307 SoRT3-17 432 58.56 1.41 MK830289 SoRT3-38 432 62.50 1.67 MK830308 SoRT3-18 432 56.94 1.32 MK830290 SoRT3-40 432 59.26 1.45 MK830309 SoRT3-19 432 59.26 1.45 MK830291 SoRT3-41 432 64.81 1.84 MK830310 SoRT3-21 432 60.65 1.54 MK830292 SoRT3-43 432 57.41 1.35 MK830311 SoRT3-22 432 62.04 1.63 MK830293 SoRT3-44 432 62.27 1.65 MK830312 SoRT3-23 432 63.66 1.75 MK830294 SoRT3-45 432 59.72 1.48 MK830313 SoRT3-24 432 63.43 1.73 MK830295 SoRT3-46 432 58.80 1.43 MK830314 SoRT3-25 432 62.73 1.68 MK830296 SoRT3-47 432 62.73 1.68 MK830315 SoRT3-26 430 57.91 1.38 MK830297 SoRT3-48 432 62.50 1.67 MK830316 SoRT3-27 432 61.34 1.59 MK830298 变化范围 Range 430~432 56.71~64.81 1.31~1.84 — SoRT3-28 432 63.66 1.75 MK830299 表 2 Ty3-gypsy类逆转座子RT基因蛋白质结构信息
Table 2. RT protein structure of Ty3-gypsy-like retrotransposons
家族
Family序列号
Sequence
No.α-螺旋数
α-helix
number aβ-折叠数
β-folding
number a转角数
Rotation
number b氢键数
Hydrogen
bond
numberbⅠ SoRT3-7 6 9 19 92 Ⅱ SoRT3-21 6 9 18 94 Ⅲ SoRT3-30 6 9 18 89 Ⅳ SoRT3-1 5 8 17 93 Ⅳ SoRT3-4 6 9 18 92 Ⅴ SoRT3-26 5 9 12 76 注:a数据结果是根据图6-A的蛋白质二级结构统计得出;b数据结果根据图6-B的蛋白质三级结构统计得出。
Note: data with a are based on protein secondary structure as shown on Fig. 6-A; while b based on tertiary structure as displayed on Fig. 6-B.表 3 部分其他物种植物的Ty3-gypsy类逆转座子RT基因序列信息
Table 3. Amino acid sequences of RT of Ty3-gypsy-like retrotransposons from some other plant species
科
Family物种
Species登录号
Accession No.松科 Pinaceae 白皮松 Phelipanche bungeana ABD43118.1 落叶松 Larix gmelinii BAQ22332.1 莎草科 Cyperaceae 荸荠 Eleocharis uniglumis ADF46121.1 藜科 Chenopodiaceae 菠菜 Spinacia oleracea BAB40833.1 禾本科 Poaceae 多花黑麦草 Lolium multiflorum BAB40827.1 粳稻 Oryza sativa japonica group BAB40824.1 豆科 Fabaceae 大豆 Glycine max BAB40834.1 绿豆 Vigna radiata AAT85841.1 蔷薇科 Rosaceae 果梅 Prunus mume ACU42190.1 苹果 Malus domestica ABS11079.1 仙人掌科 Cactaceae 火龙果 Hylocereus undatus AOS58468.1 菊科 Compositae 菊花 Chrysanthemum x morifolium BAB40831.1 列当科 Orobanchaceae 列当 Orobanche cernua ABD43042.1 毛茛科 Ranunculaceae 牡丹 Paeonia suffruticosa AFQ94056.1 十字花科 Brassicaceae 拟南芥 Arabidopsis thaliana BAB40828.1 桑科 Moraceae 山桑 Morus bombycis BAB40830.1 棕榈科 Palmae 油棕 Elaeis guineensis CAD45567.1 鼠李科 Anacardiaceae 枣 Ziziphus jujuba AFR43612.1 表 4 Ty3-gypsy类逆转座子RT基因序列与EST数据库比对结果
Table 4. Sequence alignment with EST database on RT of Ty3-gypsy-like retrotransposons
序列号
Sequence No.目标序列
Target sequence查询覆盖度
Query coverage/%E值
E value一致性
Identity/%SoRT3-9 GE325024.1 87 2×10−142 91.25 SoRT3-21 GE325024.1 87 1×10−165 94.96 SoRT3-22 GE325024.1 87 2×10−152 92.84 SoRT3-27 GE325024.1 85 5×10−154 93.53 SoRT3-29 GE325024.1 86 1×10−154 93.33 SoRT3-34 GE325024.1 86 6×10−98 84.27 SoRT3-9 GE325034.1 87 2×10−142 91.25 SoRT3-21 GE325034.1 87 1×10−165 94.96 SoRT3-22 GE325034.1 87 2×10−152 92.84 SoRT3-27 GE325034.1 85 5×10−154 93.53 SoRT3-29 GE325034.1 86 1×10−154 93.33 SoRT3-34 GE325034.1 86 6×10−98 84.27 SoRT3-23 CA177106.1 99 8×10−157 90.26 SoRT3-35 CA177106.1 99 2×10−148 89.10 SoRT3-36 CA177106.1 97 3×10−141 88.57 SoRT3-41 CA177106.1 99 2×10−148 89.10 SoRT3-23 CA203336.1 97 1×10−140 88.15 SoRT3-35 CA203336.1 97 1×10−130 86.76 SoRT3-41 CA203336.1 97 5×10−129 86.49 -
[1] WAUGH R, MCLEAN K, FLAVELL A J, et al. Genetic distribution of Bare-1-like retrotransposable elements in the barley genome revealed by sequence-specific amplification polymorphisms (S-SAP) [J]. Molecular and General Genetics, 1997, 253(6): 687−694. doi: 10.1007/s004380050372 [2] KALENDAR R, GROB T, REGINA M, et al. IRAP and REMAP: Two new retrotransposon-based DNA fingerprinting techniques [J]. Theoretical and Applied Genetics, 1999, 98(5): 704−711. doi: 10.1007/s001220051124 [3] FLAVELL A J, KNOX M R, PEARCE S R, et al. Retrotransposon-based insertion polymorphisms (RBIP) for high throughput marker analysis [J]. The Plant Journal, 1998, 16(5): 643−650. doi: 10.1046/j.1365-313x.1998.00334.x [4] 吴子莺, 杨善, 钱旺, 等. 甘蔗属大茎野生种Ty1-copia反转录转座子逆转录酶序列克隆与特点分析 [J]. 植物遗传资源学报, 2020, 21(2):466−476.WU Z Y, YANG S, QIAN W, et al. Cloning and characterisation of reverse transcriptase sequences of Ty1-copia retrotransposon in Saccharum robustum [J]. Journal of Plant Genetic Resources, 2020, 21(2): 466−476.(in Chinese) [5] RAZA S, ANJUM S, QAMARUNISA S, et al. Genome analysis of sugarcane (cultivar BL4) to investigate transposable elements [J]. Pakistan Journal of Biochemistry and Molecular Biology, 2011, 44(2): 68−72. [6] ROSSI M, ARAUJO P G, VAN SLUYS M A. Survey of transposable elements in sugarcane expressed sequence tags (ESTs) [J]. Genetics and Molecular Biology, 2001, 24(1/2/3/4): 147−154. [7] ROSSI M, ARAUJO P G, JESUS E M, et al. Comparative analysis of Mutator -like transposases in sugarcane [J]. Molecular Genetics and Genomics, 2004, 272(2): 194−203. doi: 10.1007/s00438-004-1036-2 [8] ARAUJO P G, ROSSI M, JESUS E M, et al. Transcriptionally active transposable elements in recent hybrid sugarcane [J]. The Plant Journal, 2005, 44(5): 707−717. doi: 10.1111/j.1365-313X.2005.02579.x [9] DE JESUS E M, OCHOA CRUZ E A, CRUZ G M Q, et al. Diversification of hAT transposase paralogues in the sugarcane genome [J]. Molecular Genetics and Genomics, 2012, 287(3): 205−219. doi: 10.1007/s00438-011-0670-8 [10] NAKAYAMA S. Inter-MITE polymorphisms of a newly identified MITE show relationships among sugarcane (Saccharum) species [J]. Genetic Resources and Crop Evolution, 2012, 59(7): 1389−1396. doi: 10.1007/s10722-011-9766-6 [11] ZHANG J S, SHARMA A, YU Q Y, et al. Comparative structural analysis of Bru1 region homeologs in Saccharum spontaneum and S. officinarum [J]. BMC Genomics, 2016, 17: 446. doi: 10.1186/s12864-016-2817-9 [12] ZHANG J S, ZHANG X T, TANG H B, et al. Allele-defined genome of the autopolyploid sugarcane Saccharum spontaneum L [J]. Nature Genetics, 2018, 50(11): 1565−1573. doi: 10.1038/s41588-018-0237-2 [13] 刘俊仙, 熊发前, 刘菁, 等. 用于克隆及分子标记分析的甘蔗高质量基因组DNA提取方法 [J]. 分子植物育种, 2019, 17(2):545−552.LIU J X, XIONG F Q, LIU J, et al. High quality sugarcane DNA extraction methods for cloning and molecular marker analysis [J]. Molecular Plant Breeding, 2019, 17(2): 545−552.(in Chinese) [14] KUMEKAWA N, OHTSUBO E, OHTSUBO H. Identification and phylogenetic analysis of Gypsy-type retrotransposons in the plant kingdom [J]. Genes & Genetic Systems, 1999, 74(6): 299−307. [15] 熊发前, 刘俊仙, 刘菁, 等. 花生DNA的五种改良CTAB提取方法的比较分析及其应用 [J]. 分子植物育种, 2019, 17(7):2207−2216.XIONG F Q, LIU J X, LIU J, et al. Comparative analysis and application of five improved CTAB extraction methods for peanut DNA [J]. Molecular Plant Breeding, 2019, 17(7): 2207−2216.(in Chinese) [16] 阳太亿, 刘俊仙, 刘菁, 等. 四倍体野生种花生Ty1-copia类逆转座子逆转录酶基因的克隆与分析 [J]. 山东农业科学, 2019, 51(9):9−20.YANG T Y, LIU J X, LIU J, et al. Cloning and analysis of reverse transcriptase of Ty1-copia-like retrotransposons in Arachis monticola [J]. Shandong Agricultural Sciences, 2019, 51(9): 9−20.(in Chinese) [17] 王庆竹, 李慧平, 文晓鹏, 等. 桂花LTR类反转录转座子RT序列的克隆及分析 [J]. 园艺学报, 2018, 45(2):309−320.WANG Q Z, LI H P, WEN X P, et al. Cloning and analysis of reverse transcriptase of LTR retrotransposons in Osmanthus fragrans [J]. Acta Horticulturae Sinica, 2018, 45(2): 309−320.(in Chinese) [18] 侯小改, 郭大龙, 黄燕梅, 等. 牡丹Ty3-gypsy类反转录转座子反转录酶序列的克隆及分析 [J]. 园艺学报, 2013, 40(1):98−106.HOU X G, GUO D L, HUANG Y M, et al. Cloning and analysis of reverse trancriptase of Ty3-Gypsy-like retrotransposons in tree peony(Paeonia) [J]. Acta Horticulturae Sinica, 2013, 40(1): 98−106.(in Chinese) [19] 彭磊, 吴艳, 刘小翠, 等. 火龙果Ty3-gypsy类反转录转座子反转录酶序列的克隆及分析 [J]. 果树学报, 2017, 34(2):186−195.PENG L, WU Y, LIU X C, et al. Cloning and analysis of reverse transcriptase of Ty3-Gypsy retrotransposon in Hylocereus undatus [J]. Journal of Fruit Science, 2017, 34(2): 186−195.(in Chinese) [20] 张文波, 陈凌, 李雪辉, 等. 兴安落叶松Ty3-gypsy类逆转座子逆转录酶的多样性分析 [J]. 分子植物育种, 2016, 14(5):1098−1106.ZHANG W B, CHEN L, LI X H, et al. Sequence diversity analysis of reverse transcriptases of Ty3-Gypsy-like retrotransposons in Larix gmelinii [J]. Molecular Plant Breeding, 2016, 14(5): 1098−1106.(in Chinese) [21] 白杨, 林晓飞, 张文波. 杂交构树Ty3-gypsy类逆转座子逆转录酶序列的多样性分析 [J]. 分子植物育种, 2018, 16(22):7429−7437.BAI Y, LIN X F, ZHANG W B. Diversity analysis of Ty3-Gypsy-like retrotransposon reverse transposases in Broussonetia papyrifera L. vent [J]. Molecular Plant Breeding, 2018, 16(22): 7429−7437.(in Chinese) [22] TANG Y M, MA Y Z, LI L C, et al. Identification and characterization of reverse transcriptase domain of transcriptionally active retrotransposons in wheat genomes [J]. Journal of Integrative Plant Biology, 2005, 47(5): 604−612. doi: 10.1111/j.1744-7909.2005.00055.x -






 下载:
下载: