2. 云南省都市特色农业工程技术研究中心, 云南 昆明 650214;
3. 中国科学院昆明动物研究所, 云南 昆明 650223;
4. 昆明学院生命科学与技术系, 云南 昆明 650214
2. Engineering Research Center for Characteristics Agriculture of Yunnan Province, Kunming, Yunnan 650214, China;
3. Kunming Institute of Zoology, the Chinese Academy of Sciences, Kunming, Yunnan 650223, China;
4. Life Science and Technology Department, Kunming University, Kunming, Yunnan 650214, China
铁皮石斛Dendrobium candidum,为兰科多年生附生草本植物。生于海拔达1 600 m的山地半阴湿的岩石上,喜温暖湿润气候和半阴半阳的环境,不耐寒。石斛可分为黄草、金钗、马鞭等数十种,铁皮石斛为石斛之极品,它因表皮呈铁绿色而得名[1]。现代研究表明,其含石斛碱、石斛胺、石斛次碱、石斛星碱、石斛因碱、6-羟石斛星碱、3-羟基-2-氧石斛碱,还含有黏液质、淀粉等[2]。故有促进肠胃蠕动,增加胃液分泌,助消化,降血压,降血糖,升高白细胞,增强机体免疫力等作用。此外,石斛所含生物碱还有抗菌、止痛、退热等作用,并被认为有抗癌效能[3]。据我国有关科研人员分析,金钗石斛的挥发油含有53种化学成分,主要有柏泪醇(50.46%)、紫罗兰酮、十八碳二烯-9,12-酸等[4]。
内生真菌是在宿主植物体内生存并完成生活周期的真菌,几乎所有活体植物内均有内生真菌的存在。内生真菌在石斛属植物种子萌发[5]、抵御病原菌侵染[6]、总生物碱及多糖积累[7]、促进幼苗生长中起着重要的作用[8],同时内生真菌也是生物活性物质筛选的重要来源[9]。
传统的真菌分类鉴定主要是按照真菌的形态、生长以及生理生化等特征对真菌进行分类[10]。然而真菌的种类繁多,个体多态性不明显,而且其生长、生理生化特征也会随着环境的变化而不稳定[11]。因此,采用传统的方法对真菌进行正确的分类存在较大的困难。随着分子生物学技术的发展,核酸序列分析已被广泛应用于真菌分类鉴定中,目前常用的技术包括18S rDNA[12]、内转录间区(ITS,Internal Transcribed Spacer)。ITS常用作微生物分类研究的包含ITS1和ITS2两种[13]。ITS1位于真核生物核糖体rDNA序列的18S和5.8S之间,ITS2位于真核生物核糖体 rDNA序列5.8S和28S之间[14]。由于不需要加入成熟核糖体,因此在进化过程中能够承受更多的变异,其进化速率为18S rDNA的10倍,属于中度保守的区域,利用它可研究种及种以下的分类阶元[15]。另外,也可通过选择引物同时扩增18S rDNA和ITs,通过分析18S rDNA序列,先在较高级别上确定样品的归属,然后根据ITS 序列,将真菌归类到种或亚种水平[16]。以往454测序平台以读长优势广泛应用于18S rDNA测序领域,随着MiSeq平台双端PE250和PE300测序策略的发展,读长不断增长,使得基于MiSeq测序研究物种多样性的准确性大幅度提高[17]。利用MiSeq平台对ITS1或ITS2区进行测序,具有测序深度高、利于鉴定低丰度群落物种以及费用低的特点,已成为研究真菌群落多样性的首选之策[18]。
目前已报道的关于研究石斛内生真菌多样性研究均是采用传统分离培养方法进行的,被鉴定的内生真菌种类归属于担子菌门、半知菌门和子囊菌门中几十个属[1]。而许多研究已经证实,通过传统的分离培养方法鉴定的微生物只占环境生物总数的0.1%~10%[19, 20],所以通过传统培养方法并不能真实反映出石斛内生真菌多样性。目前采用非培养方法对石斛内生真菌的研究尚未见报道,因此本研究采用近年来兴起的Illumina MiSeq第二代测序技术对铁皮石斛内生真菌种类组成进行研究,相较于传统的纯培养方法及以16S rDNA为基础的非培养方法,能够产生测序覆盖深度更大的数据量,从而对铁皮石斛内生真菌的种类进行全面而准确的调查,为丰富植物微生态学理论及基因工程菌的研究和应用奠定基础。
1 材料与方法 1.1 样品采集2014年8月于云南省普洱市景东彝族自治县野生铁皮石斛产地采集20株无明显病害的野生铁皮石斛,随机摘取叶片,均匀混合后放入无菌样品袋中,低温保鲜,于24 h内进行表面消毒处理,样品名为SY。
1.2 表面消毒将铁皮石斛叶片用无菌水冲洗干净,再用75%(体积分数)的乙醇浸泡1 min,无菌水冲洗3次,用无菌滤纸吸干表面水分,表面消毒效果检验参照文献[21]的方法。
1.3 总DNA提取按参考文献[22]的方法提取叶片总DNA,并用8 g·L-1的琼脂糖凝胶电泳检查DNA的纯度和浓度,取适量的样品于离心管中,使用无菌水稀释样品至1 mg·L-1。
1.4 ITS2区的PCR扩增以稀释后的基因组DNA为模板,使用带Barcode的ITS2区特异引物3F(5′-TTACT GGCATCGATGAAGAACGCAGC-3′)和4R(5′-GGTGCTTCCTCCGCTTATTGATATGC-3′),使用高效和高保真酶(New England Biolabs公司的Phusion High-Fidelity PCR Master Mix with GC Buffer)进行PCR,确保扩增效率和准确性。
1.5 PCR产物的纯化PCR产物使用20 g·L-1浓度的琼脂糖凝胶进行电泳检测,目的条带使用Qiagen公司提供的胶回收试剂盒回收产物。
1.6 文库构建和上机测序用TruSeq® DNA PCR-Free Sample Preparation Kit建库试剂盒进行文库构建,构建好的文库经过Qubit和QPCR定量,文库合格后,使用Miseq 进行上机测序[23, 24, 25]。测序委托北京诺禾致源生物信息科技有限公司完成。
1.7 生物信息学分析测序得到的原始数据存在一定比例的干扰数据,为了使信息分析的结果更加准确、可靠,首先对原始数据用Flash软件拼接、Qiime软件过滤、Uchime Algorithm软件去除嵌合体后,得到有效数据(Effective tags)[26],然后用Uparse软件对有效数据在97%水平上进行操作分类单元(Operational taxonomic unit,OTU)聚类,并利用Greengene数据库进行物种注释[27],利用Mothur软件做稀释曲线分析,通过对OTUs 进行丰度和α-多样性分析,可以得到微生物群落结构组成[28, 29]。
2 结果与分析 2.1 序列长度分布所测得的48 199条双末端读长序列(PE,Paired-end reads)长度分布在294~414 bp范围内,长度为384 bp的序列最多,有41 181条。从序列长度的分布来看,与ITS2区序列长度大致吻合。
2.2 OTUs数目统计及物种注释分析共获得41 361条序列(Tags,过滤后得到的拼接序列数),可分为33个操作分类单元(OTUs,97%的序列相似性,下同),包括452条低频序列和40 909条可以获得注释的序列(图 1)。
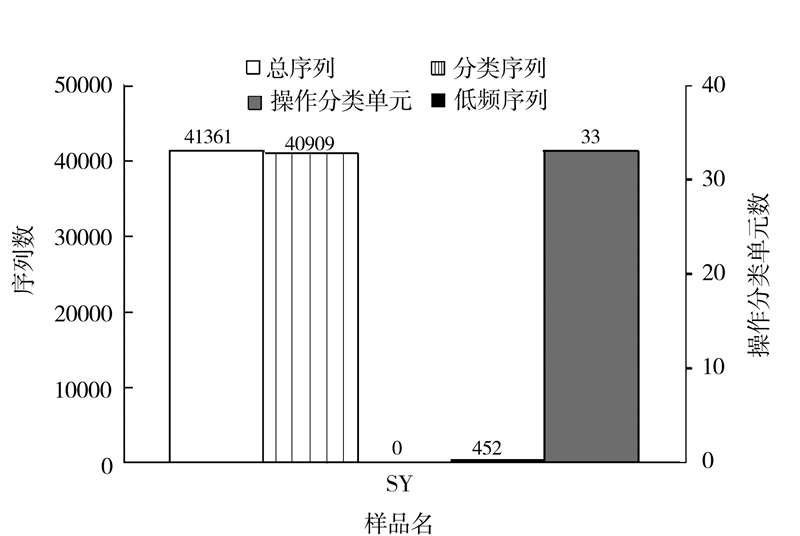
|
图 1 样品SY的序列和操作分类单元数目统计 Fig.1 Statistics on Tags and OTUs of Sample SY |
特定特种分类树如图 2所示,从门的分类水平来看,铁皮石斛内生真菌主要分布在子囊菌门(Ascomycota,99.97%),仅有少量分布于担子菌门(Basidiomycota,0.03%);从纲的分类水平来看,铁皮石斛内生真菌主要分布在散囊菌纲(Eurotiomycetes,99.76%),少量分布于座囊菌纲(Acidobacteria,0.06%)、茶渍纲(Lecanoromycetesa,0.06%)、锤舌菌纲(Leotiomycetes,0.04%)、粪壳菌纲(Sordariomycetes,0.045%)、伞菌纲(Agaricomycetes,0.03%),其次还有0.01%的序列经Greengene数据库比对,在纲的分类水平上尚无物种注释信息,推测可能是潜在的未知种类。由此可见,铁皮石斛内生真菌99%以上分布于子囊菌门的散囊菌纲,其他类群的序列所占比例均小于1%。

|
图 2 样品SY中特定物种分类树 注:数字表示该分类所占比例,“Un-s-”代表序列在该分类水平上尚无物种注释信息。 Fig.2 Classification tree of specific species from Sample S |
从目的分类水平来看,铁皮石斛内生真菌主要分布于散囊菌目(Eurotiales,99.69%);从科的分类水平来看,铁皮石斛内生真菌主要分布于发菌科(Trichocomaceae,99.69%);从属的分类水平来看,铁皮石斛内生真菌主要分布于裸胞壳属(Emericella,99.57%);从种的分类水平来看,铁皮石斛内生真菌主要为构巢裸壳孢菌(Emericella nidulans,99.57%),由此可见,构巢裸壳孢菌为铁皮石斛内生真菌的优势种群。
2.4 样品复杂度分析从稀释曲线(图 3)来看,随着测序数量的增加,稀释曲线斜率逐渐降低,趋向平缓但未进入平台期,说明再增加测序数量也只会产生少量新的OTUs;从Chao1指数曲线(图 4)来看,Chao1指数在0~15 000的测序数量范围内增幅最大,在15 000以后曲线斜率下降;从Shannon指数曲线来看(图 5),Shannon指数在0~5 000的测序数量范围内增幅最大,在5 000以后曲线斜率下降。由此可见,3条曲线在测序数量超过一定数量后都趋近平缓,说明测序数据量足够大,可以反映样品中绝大多数的微生物信息。

|
图 3 稀释曲线 Fig.3 Rarefaction curve |

|
图 4 Chao1 指数曲线 Fig.4 Chao1 index curve |

|
图 5 Shannon指数曲线 Fig.5 Shannon index curve |
本研究首次将近几年迅速发展并成为主流的Illumina MiSeq第2代测序技术应用于植物内生真菌研究,克服了传统分子生物学方法存在的通量低的缺陷,从基因组的水平上来解析微生物群落结构,突破了很多厌氧内生微生物尚不能被分离培养的技术瓶颈[19, 20],相较于传统的纯培养方法及以16S rDNA为基础的非培养方法,覆盖了整个内生微生物群落的信息,可以检测到以往没有发现的但同样扮演者重要角色的低丰度植物内生真菌种类,例如:座囊菌纲(Acidobacteria,0.06%)、茶渍纲(Lecanoromycetesα,0.06%)、锤舌菌纲(Leotiomycetes,0.04%)等,丰富了植物内生真菌资源。本研究证实了铁皮石斛中确实存在内生真菌,显示出高通量技术在植物内生微生物研究中的可行性及明显优势,相较于传统分子生物学研究结果的片面性,高通量测序技术准确地反映植物内生微生物中高丰度菌种的组成和真实比例。
朱琳等应用Illumina Miseq高通量测序技术对文冠果根部内生真菌的ITS1区进行了测序[30],发现在门的分类水平上,内生真菌主要分布于子囊菌门(27.0%)、担子菌门(7.5%);在纲的分类水平上,主要分布于座囊菌纲(26.0%)、粪壳菌亚纲(16.0%)、银耳纲(7.2%)、散囊菌纲(2.8%);在目的水平上,主要分布于格孢菌目(26.0%)、肉座菌目(14.0%)、散囊菌目(2.7%)、银耳目(0.6%)。由此可见,在门的分类水平上,文冠果根部内生真菌的种类分布与本研究得出的结果一致,在纲的分类水平上,本研究同样检测到座囊菌纲(0.06%)、粪壳菌亚纲(0.045%)和散囊菌纲(99.76%)真菌,但是所占比例不一致,未检测到银耳纲;在目的分类水平上,本研究同样检测到散囊菌目(99.69%),但是所占比例不一致,未检测到格孢菌目、肉座菌目、银耳目真菌。推测可能与宿主植物不同有关,现有研究表明,不同内生真菌对不同的植物有着特异的选择偏好性[1]。本研究发现铁皮石斛内生真菌从门的分类水平上看主要分布于子囊菌门(99.97%),这与张延威等[31]和陈玉栋等[32]采用传统分离培养方法研究的结果相一致。从属的分类水平上来看,铁皮石斛内生真菌主要分布于翘孢霉属(99.57%),这与洪群艳等[33]认为刺盘孢属为茎和叶的优势种群的结果不一致。这可能与石斛的品种、采集地、生长环境有关,有研究表明,同种植物在不同的地方,可能有不同的内生真菌[34];其次可能与研究方法不同有关,采用传统分离培养方法可能与非培养方法得出的结果不一致,例如在采用构建克隆文库的非培养方法对植物内生细菌多样性的研究中普遍认为变形菌门(Proteobacteria)为优势类群,而与培养方法得出的厚壁菌门(Firmicutes)为优势类群的结论不一致[35, 36]。这是因为培养方法分析的微生物只限于那些能够在人工培养基上生长的种类,因此通过传统培养方法得到的结果并不能真实地反映出内生微生物的多样性。从属的分类水平来看,铁皮石斛内生真菌主要为裸胞壳属的构巢裸壳孢菌(Emericella nidulans,99.57%)。现有的内生真菌多样性研究报道表明,裸胞壳属真菌曾多次被分离自毛脉酸模[37]、黄芪[38, 39]等常用中药材植物。目前关于裸胞壳属构巢裸壳孢菌(Emericella nidulans)次级代谢产物的研究较少,主要从该菌中分离得到了系列的三酮类化合物[40]和喹啉类生物碱化合物[41],未来应进一步加强对其功能和代谢的研究。
进入 21 世纪后,人类基因组计划揭开了大数据时代,各国学者对微生物多样性的研究已从形态特征分析、生理生化分析、分子生物学技术等传统研究手段向高通量测序技术分析逐渐转化。相信随着第二代测序技术的快速发展,人们对环境微生物的认识将会不断拓展,使其成为对难以培养微生物或不可培养微生物群落研究的首选技术策略,让人们直接对植物总DNA进行测序分析,从整体水平上尽可能揭示植物内生微生物的群落结构及多样性。
| [1] | 方香香,张寿文. 铁皮石斛内生真菌研究进展[J]. 中国现代中药,2014,16(5):419-422.( 3) 3) |
| [2] | 王明月,陶茜,李克艳,等. 铁皮石斛内生细菌群落结构分析[J]. 西部林业科学,2014,43(5):106-111.( 1) 1) |
| [3] | 王亚妮,王丽琨,苗宗保,等. 流苏石斛根部内生真菌多样性研究[J]. 生态科学,2014,33(4):672-679.( 1) 1) |
| [4] | 严亮,杨瑞娟,王桥美. 云南铁皮石斛内生菌的分离与鉴定[J]. 云南农业大学学报,2015,30(5):760-765.( 1) 1) |
| [5] | 侯晓强,郭顺星. 铁皮石斛促生长内生真菌的筛选与鉴定[J]. 中国中药杂志,2014,39(17):3232-3237.( 1) 1) |
| [6] | 何劲,雷帮星,宋贞富,等. 石斛内生细菌DEB-2对5种辣椒病原真菌的抑制作用[J]. 植物保护学报, 2014, 41(2):157-162.( 1) 1) |
| [7] | 杨绍周,吴毅歆,邵德林,等. 鼓槌石斛内生细菌分离、鉴定及功能分析[J]. 中国农学通报,2014,30(25):171-176.( 1) 1) |
| [8] | 徐文婷,张雅琼,董文汉,等. 石斛内生真菌固体菌剂对铁皮石斛组培苗促生作用研究[J]. 西南农业学报,2014,27(1):317-324.( 1) 1) |
| [9] | 童文君,张礼,薛庆云,等. 不同产地美花石斛内生细菌分离及促生潜力比较[J]. 植物资源与环境学报,2014,23(1):16-23.( 1) 1) |
| [10] | CHEUNG M K, AU C H, CHU K H, et al. Composition and genetic diversity of picoeukaryotes in subtropical coastal waters as revealed by 454 pyrosequencing[J]. ISME J, 2010, 4(8): 1053-1059.( 1) 1) |
| [11] | OROS-SICHLER M, GOMES N C, NEUBER G, et al.A new semi-nested PCR protocol to amplify large 18S rRNA gene fragments for PCR-DGGE analysis of soil fungal communities[J]. J Microbiol Methods, 2006, 65(1): 63-75.( 1) 1) |
| [12] | STOECK T, BEHNKE A, CHRISTEN R, et al. Massively parallel tag sequencing reveals the complexity of anaerobic marine protistan communities[J]. BMC Biol, 2009,(7): 72.( 1) 1) |
| [13] | BACHY C, DOLAN J R, LÓPEZ-GARCÍA P, et al. Accuracy of protist diversity assessments: morphology compared with cloning and direct pyrosequencing of 18S rRNA genes and ITS regions using the conspicuous tintinnid ciliates as a case study[J]. ISME J, 2012, 7(2): 244-255.( 1) 1) |
| [14] | JOHAN B P, MARTIN R, MARTIN H, et al. Improved software detection and extraction of ITS1 and ITS2 from ribosomal ITS sequences of fungi and other eukaryotes for analysis of environmental sequencing data[J]. Methods in Ecology and Evolution, 2013, 4(10): 914-919.( 1) 1) |
| [15] | FINDLEY K, OH J, YANG J, et al. Topographic diversity of fungal and bacterial communities in human skin[J]. Nature, 2013, 498(7454): 367-370.( 1) 1) |
| [16] | LUO C, TSEMENTZI D, KYRPIDES N, et al. Direct comparisons of Illumina vs. Roche 454 sequencing technologies on the same microbial community DNA sample[J]. PloS one, 2012, 7(2): e30087.( 1) 1) |
| [17] | WU L, WEN C, QIN Y, et al. Phasing amplicon sequencing on Illumina Miseq for robust environmental microbial community analysis[J]. ISME J, 2012,(15): 125.( 1) 1) |
| [18] | CAPORASO J G, LAUBER C L, WALTERS W A, et al. Ultra-high-throughput microbial community analysis on the Illumina HiSeq and MiSeq platforms[J]. ISME J, 2012, 6(8): 1621-1624.( 1) 1) |
| [19] | THOLOZAN J L, CAPPELIER J M, TISSIER J P, et al. Physiological characterization of viable-but-nonculturable Campylobacter jejuni cells[J]. Appl Environ Microbiol, 1999, 65(3): 1110-1116.( 2) 2) |
| [20] | AMMANN R R,LUDWIG W,SCHLEIFER K H. Phylogenetic identification and in situ detection of individual microbial cells without cultivation[J]. Microbiol Rev, 1995, 59(1):143-169.( 2) 2) |
| [21] | 陈泽斌,代方平,寸林江,等. 烟草内生细菌分离方法的优化研究[J]. 中国烟草学报,2014,20(1):90-95,102.( 1) 1) |
| [22] | 邱服斌. 培养方法与非培养方法对人参根内生细菌的研究[D]. 北京:首都师范大学,2007.( 1) 1) |
| [23] | CAPORASO J G,LAUBER C L,WALTERS W A,et al. Global patterns of 16S rRNA diversity at a depth of millions of sequences per sample[J]. Proceedings of the National Academy of Sciences,2011,108 (S1):4516-4522.( 1) 1) |
| [24] | YOUSSEF N,SHEIK C S,KRUMHOLZ L R,et al. Comparison of species richness estimates obtained using nearly complete fragments and simulated pyrosequencing-generated fragments in 16S rRNA gene-based environmental surveys[J]. Applied and Environmental Microbiology, 2009,75(16):5227-5236.( 1) 1) |
| [25] | HESS M,SCZYRBA A,EGAN R,et al. Metagenomic discovery of biomass-degrading genes and genomes from cow rumen[J]. Science,2011,331(6016):463-467.( 1) 1) |
| [26] | EDGAR R C,HAAS B J,CLEMENTE J C,et al. UCHIME improves sensitivity and speed of chimera detection[J]. Bioinformatics,2011,27(16):2194-2200.( 1) 1) |
| [27] | EDGAR R C. UPARSE:highly accurate OTU sequences from microbial amplicon reads[J]. Nature Methods,2013,10(10):996-998.( 1) 1) |
| [28] | WANG Q,GARRITY G M,TIEDJE J M,et al. Naive Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J]. Applied and Environmental Microbiology,2007,73(16):5261-5267.( 1) 1) |
| [29] | DESANTIS T Z,HUGENHOLTZ P,LARSEN N,et al. Greengenes,a chimera-checked 16S rRNA gene database and workbench compatible with ARB[J]. Applied and Environmental Microbiology,2006,72(7):5069-5072.( 1) 1) |
| [30] | 朱琳,黄建,陈天阳,等. 文冠果人工林根际土壤真菌和根系内生真菌群落多样性[J]. 东北林业大学学报,2015,43(5):105-111.( 1) 1) |
| [31] | 张廷威,韩燕峰. 石斛内生真菌多样性的初步研究[J]. 贵州教育学院学报,2009,20(6):9-12.( 1) 1) |
| [32] | 陈玉栋,李文锦,雷玲,等.铁皮石斛内生真菌的分离与鉴定[J].信阳师范学院学报,2009,22(2):236-238.( 1) 1) |
| [33] | 洪群艳,董文汉,徐文婷,等.几种云南野生石斛内生真菌的鉴定及分布[J]. 生物技术进展,2012,2(3):190-194.( 1) 1) |
| [34] | ESPINOSA-GARCIA F J,LANGENHEIM J H. The leaf fungal endophytic community of a coastal redwood population diversity and spatial patterns[J]. New Phytol,1990,116:89-97.( 1) 1) |
| [35] | 孙磊. 非培养方法和培养方法对水稻内生细菌和根结合细菌的研究[D]. 北京:首都师范大学, 2006.( 1) 1) |
| [36] | 程晓燕, 李文军, 王芸, 等. 新疆野生胀果甘草内生细菌多样性的非培养初步分析[J]. 微生物学报, 2009, 49(6): 718-725.( 1) 1) |
| [37] | 胡凤. 毛脉酸模内生真菌化学成分的分析及其分子鉴定[D]. 哈尔滨:黑龙江中医药大学,2009.( 1) 1) |
| [38] | 马伟,贾艳姝,李娜,等. 植物黄芪根内生真菌的分离[J]. 东北林业大学学报,2012,40(4):62-63,116.( 1) 1) |
| [39] | 孔祥军. 蒙古黄芪内生真菌分离鉴定及活性成分初步研究[D]. 哈尔滨:黑龙江中医药大学, 2011.( 1) 1) |
| [40] | 赵玲玲. 海洋真菌中新型抗肿瘤活性物质的研究[D]. 上海:复旦大学,2008.( 1) 1) |
| [41] | 孙新华. 三株滨海来源植物内生真菌次级代谢产物及其活性研究[D]. 青岛:中国海洋大学,2014.( 1) 1) |
 2015, Vol. 30
2015, Vol. 30
